Application Note ClonePixテクノロジーによる
GPCR細胞株の効率的スクリーニング
- 内因性レベルでGPCRターゲットタンパク質を哺乳類発現システムで迅速に評価します
- 最適なプロデューサーを見つける可能性を高めます
- セルラインや抗体開発の時間を短縮 ― リミティングダイリューションを回避します
PDF版(英語)
ClonePix™ 2システムは、哺乳類細胞株におけるGPCRターゲットタンパク質の発現レベルを迅速に評価する新しい方法を提供します。
背景
哺乳類細胞におけるGPCRの内因性発現は通常非常に低く、細胞あたり3,000コピーを超えることはほとんどありません。これらの内因性レベルは適切な受容体機能を維持するには十分ですが、GPCR創薬においては課題となります。ほとんどのスクリーニングアッセイでは、細胞表面に提示される機能的GPCRの濃度がはるかに高いことが求められます。たとえば、構造解析、低分子薬設計、ネイティブGPCRターゲットに対する機能抗体の作製には、内因性レベルよりも桁違いに高い発現量が必要です。 「低次」生物で発現系を構築しようとする試みは、折り畳みの非効率性(細菌)、低収率(酵母)、翻訳後修飾の不適切さ(バキュロウイルス)により、限定的な成功にとどまっています。これらの課題は、創薬を支援するために必要な高レベルGPCRタンパク質発現を提供できる哺乳類発現システムへの市場ニーズを高めています。
トランスフェクトされた細胞プールから高発現GPCRクローンを探索・選択することは、セルライン開発において困難です。ClonePix 2システムの技術は、10,000クローンを3週間でスクリーニングできる実績あるワンステップ法を提供し、解析可能な細胞プールが大幅に増えることで最適なプロデューサーを見つける確率を高めます。ClonePix 2システムは、白色光と蛍光画像をin situで利用し、細胞表面発現に限定された内因性レベルのタンパク質を検出する感度を備えています。
方法
内因性Gタンパク質共役ムスカリン1コリン作動性受容体(GPCR-M1)を発現するトランスフェクトCHO-M1細胞株を選択し、ClonePix 2システムで細胞表面発現タンパク質を検出する実現可能性を示しました。CHO-M1発現クローンは、PE(フィコエリスリン)標識抗M1抗体を用いてスクリーニングし、蛍光強度に基づいて選択し、ClonePix 2システムでピッキングしました。親株CHO-K1(ワイルドタイプ)はネガティブコントロールとして使用しました。ClonePix 2システムでピックしたコロニーは、ラベルフリーで細胞増殖を客観的に評価するCloneSelect™ Imagerで増殖をモニターしました。ClonePix 2システムで単離したクローンの機能検証は、モレキュラーデバイスのFLIPR® TetraハイスループットセルベーススクリーニングシステムとFLIPR® Calcium 6アッセイキットを用いて実施しました。FLIPR Tetraシステムは、GPCR活性化後の細胞内カルシウム応答変化を蛍光カルシウム感受性レポーターダイで検出する創薬における標準的なハイスループット機能アッセイを提供します。
材料
- セルライン 内因性Gタンパク質共役型ムスカリン性アセチルコリン受容体M1(M1 WT3)を発現するCHO-M1細胞株(ATCC。ネガティブコントロールとして親細胞CHO-K1を使用。
- 抗体: ウサギ抗ChRM1- PE標識、ポリクローナル(Bioss);ウサギ抗CHRM1ポリクローナルAbs、ノンジュゲート(Bioss);ヤギ抗ウサギIgG- PE標識、ポリクローナル(Life Technologies)。
- 培地: ハムF12培地(Corning);CloneMedia-CHO-S(モレキュラーデバイス);ウシ胎児血清(Hyclone)、1% Pen/Strep/グルタミン(Life Technologies)、450μg/mL Geneticin、G418(Life Technologies)。
- アッセイバッファー: 10X HBSS(Life Technologies)を注射用滅菌水(Irvine Scientific)で希釈し、20mM HEPES(Life Technologies)を加える。pHを7.4に調整する。
- FLIPR® Calcium 6 Assay Kits(モレキュラーデバイス、カタログ番号R8190)。
- プレーティング: 6ウェル非TC処理透明底プレート(Greiner Bio-One);96ウェル透明F底プレート(Greiner Bio-One);384ウェル黒壁無菌TC処理透明底プレート(Corning)。
- M1 AChRアゴニスト: 塩化カルバモイルコリンまたはカルバコール(Sigma)
- 浮遊細胞株、接着細胞株ともに、以下のような複数のアプリケーションに対応:
⚪︎目的の抗体を分泌するハイブリドーマのスクリーニングと選択
⚪︎細胞株開発
CloneSelect Imager
- 細胞のラベルフリー白色光イメージング
- 細胞増殖の対物レンズによる定量評価
- シンプルでユーザーフレンドリーなソフトウェアインターフェース
- 96ウェルプレートの各ウェルにおける成長速度を正確に測定
- 以下のような複数のアプリケーションをサポート
◦細胞増殖のモニタリング
◦モノクローナリティの検証
FLIPR Tetraシステム
- 標準EMCCD蛍光またはオプションのICCD蛍光および発光検出
- ユーザー設定可能な96、384、1536ウェルピペッター
- ユーザー交換可能なセル懸濁液オプション
- ユニークで設定可能な励起光学系による色素能力の拡張
- 直感的でユーザーフレンドリーなソフトウェアインターフェース
- TetraCycler内部プレートハンドラー、ロボットによるスループット向上
半固体培地への細胞播種
CHO-M1およびCHO-K1細胞株:両細胞株をCloneMedia CHOに最終濃度1,000 cells/wellで播種し、37°Cで8~10日間インキュベートして離散コロニーを形成させました。M1 GPCR発現レベルの違いを確認するため、新旧パッセージのCHO-M1細胞を播種しました。
直接標識法: PE標識抗ムスカリン1受容体(CHRM1)抗体を細胞とともに半固体培地に直接添加しました。抗体なしのコントロールウェルも播種しました。
二重標識法: 直接標識抗体が利用できない場合を想定し、一次抗体と二次抗体の使用を検証しました。非結合ウサギ抗ムスカリン抗体とPE標識抗ウサギポリクローナル抗体を細胞とともに半固体培地に添加しました。抗体なしのコントロールウェルと二次抗体のみを含むウェルも播種しました。
ClonePix 2システム概要
- 哺乳類クローンのスクリーニングと選択を自動化
- 白色光および蛍光でイメージング
- 透過照明により低コントラストコロニー(接着単層など)のイメージングをサポート
- 最大5種類の励起/蛍光フィルターペアをソフトウェアで切り替え
- ユーザー定義基準でコロニーをランク付け
- ターゲットコロニーを識別し、96ウェルプレートにピッキング
ClonePix 2システムによるGPCR(M1)発現クローンのイメージングとピッキング
陽性(CHO-M1)および陰性(CHO-K1)細胞をClonePix 2システムでイメージングし、ピッキングしました。明視野で各コロニーの位置と形態を確認し、蛍光でM1の高発現クローンを識別しました。PE標識プローブにはCy5チャンネルを使用し、露光時間は6,000 msでした。
CHO-M1陽性細胞は、直接標識抗体および二重抗体法の両方で、ClonePix 2システムで記録された蛍光信号の範囲を示しました。これはM1 GPCRの発現レベルの違いに対応します(図1)。予想どおり、親株CHO-K1は蛍光信号を示しませんでした。

図1. ClonePix 2システムによるCHO-M1細胞の検出 ClonePix 2システムは、CHO-M1細胞株で蛍光強度の多様なレベルを示し、GPCR M1タンパク質の発現レベルの違いを識別できることを示しています。ソフトウェアで認識されたコロニーは、明視野チャンネルでカラー枠で囲まれています。蛍光強度は、コロニーの物理的位置に基づいて算出されます。
細胞は、形態と蛍光強度に基づいてコロニーを識別・分類するためにグループ化されました。ピッキング対象コロニーの許容形態は、サイズ、形状、および隣接コロニーとの距離に基づきました。形態的に理想的なコロニーは内部蛍光強度でランク付けされ、4つの蛍光グループにゲート分けされました:High、Medium、CHO-K1 neg、Ungated(図2)。CHO-K1 negグループは背景信号の強度で定義され、このグループ内のすべてのコロニーは対応するネガティブコントロールウェル由来であることが確認されました。Ungatedコロニーは、背景より高いが低レベルの信号を持つコロニーを指します。
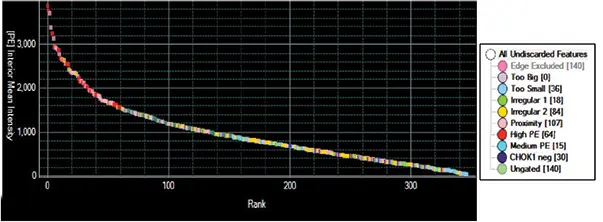
図2. CHO-M1発現の蛍光ランク付け クローンはランク付けされ、形態と内部平均蛍光強度に基づいてグループ化されました。
直接標識抗体と二重抗体法(図3)は、陽性サンプル(M1発現)で蛍光信号を示し、親株では信号が検出されませんでした(図4)。予想どおり、二重標識法では一次抗体と二次抗体の結合によりPEチャンネルで背景信号が増加しました。しかし、ClonePix 2システムは明視野でコロニーを検出し、その内部の蛍光信号を確認します。PEチャンネルで認識されても明視野で確認できないオブジェクトはコロニーとは見なされません。このため、ClonePix 2システムは背景信号から真のコロニーを識別する利点を提供します。

図3. ClonePix 2システムでイメージングしたCHO-M1陽性細胞 ClonePix 2システムによる蛍光画像解析とカスタムクローン選択。GPCR M1陽性クローンの画像を示します。直接標識抗体および二重標識抗体法によるM1発現コロニーの選択例を示します。

図4. CHO-M1陰性細胞 親株CHO-K1細胞株(ネガティブコントロール)、PE蛍光および明視野画像を示します。
ClonePix 2システムでピックしたコロニーは、CHO-M1クローン用にHam’s F12培地+10%FBS+G418を含む96ウェルプレートに、CHO-K1クローン用にはG418なしの同条件培地に移しました。これらの96ウェルプレートはCloneSelect Imagerで撮像し、移送と増殖を確認しました(図5)。細胞は1週間培養後、CloneSelect Imagerで増殖を確認しました(図5および図6)。その後、細胞は384ウェルプレートに移し、FLIPR TetraシステムとFLIPR Calcium 6アッセイを用いて発現の機能検証を行いました。
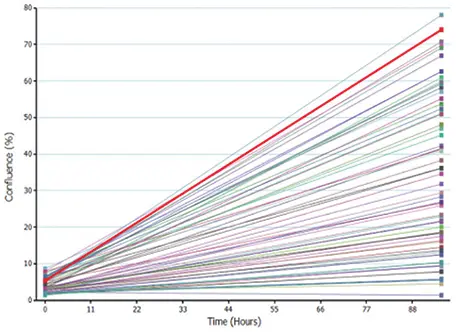
図5. CHO-M1細胞の増殖曲線 CloneSelect Imagerによる増殖曲線の生成。ピックした細胞の増殖を示します。
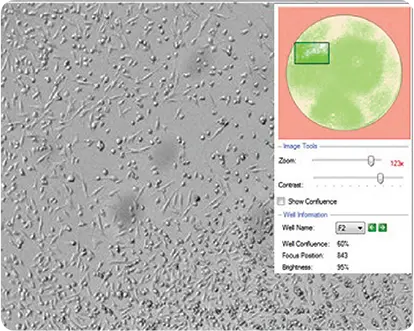
図6. CloneSelect Imagerでイメージングした細胞 CloneSelect Imagerで取得したCHO-M1細胞の画像。コンフルエンスはソフトウェアで自動計算されます。
FLIPR TetraシステムによるM1 GPCR発現の検証
細胞調製
ClonePix 2システムで選択した細胞を、384ウェルプレートに1ウェルあたり5,000細胞(培地25 µL)で播種し、37°C、95%湿度、5%CO₂で一晩培養しました。これらの細胞はClonePixシステム技術に基づき、High、Medium、Low、No M1発現の4グループに分類されました。
カルシウム感受性蛍光色素のロード
細胞プレートをインキュベーターから取り出し、室温に平衡化後、培地を除去せずにFLIPR Calcium 6 Dyeを25 µL添加しました。プレートは37°Cで2時間、2.5 mMプロベネシド存在下でインキュベートし、有機アニオントランスポーターを阻害しました。その後、蛍光測定まで室温で保持しました。
FLIPR Tetraシステム
インキュベーション後、プレートをFLIPR Tetraシステムのプラットフォームに設置し、ICCDカメラで蛍光カルシウム信号を取得しました。パラメータは以下のとおりです:
- 露光時間:0.53秒
- 励起LED:470–495 nm
- 蛍光フィルター:515–575 nm
- LED強度:80
FLIPR Calcium 6アッセイ
蛍光測定は1秒ごとに行い、カルバコール40 nM添加前後でそれぞれ10回および60回測定しました。カルバコールの初期濃度は最終濃度の5倍に設定し、添加量は12.5 µL、分注速度は20 µL/秒でした。
FLIPR Calcium 6アッセイによるCHO-M1およびCHO-K1クローンのバリデーション
膜結合Gタンパク質共役ムスカリン受容体の発現レベルは、PE標識抗M1抗体を用いて新旧パッセージのCHO-M1細胞で評価しました。古いパッセージではM1 GPCR発現レベルが新しいパッセージより有意に低いことが予想されました。親株CHO-K1細胞もネガティブコントロールとして含めました。
GPCRがリガンドで活性化されると、受容体構造が変化し、細胞内でGタンパク質が活性化されます。活性化されたGタンパク質は、カルシウムを含む細胞内メッセンジャーのカスケードを誘導します。
ClonePix 2システムでピックした各グループの細胞は、FLIPR Tetraシステムで機能活性を評価しました。カルシウム感受性蛍光色素(FLIPR Calcium 6アッセイキット)を用いて、カルバコール40 nMによるGタンパク質共役IP₃感受性経路の活性化を介した細胞質カルシウム変化を測定しました。カルバコール40 nMは、過去の濃度反応曲線に基づくEC₈₀濃度です。
CHO-M1高蛍光クローンでは、カルバコールにより背景から4倍の蛍光増加が見られました(図7A)。CHO-M1中・低蛍光クローンでは、それぞれ4倍および2倍の増加が確認されました(図7B)。CHO-K1陰性グループでは、蛍光変化は認められませんでした(図7C)。各グループは別々の384ウェルプレートに播種されていたため、ベースライン蛍光強度の変化は、カルバコール添加前に各プレートの背景蛍光値に正規化しました。
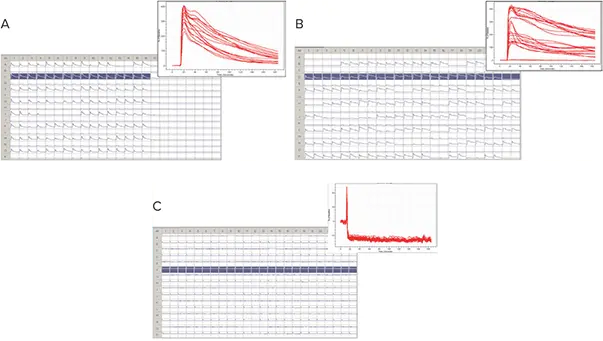
図7. FLIPR Tetraシステムによる解析 FLIPR Tetraシステムは、蛍光カルシウム感受性レポーターダイ(FLIPR Calcium 6アッセイキット)を用いて細胞内カルシウム変化を評価する、創薬における標準的なハイスループット機能セルベースアッセイを実行します。結果は以下のとおりです:(A)CHO-M1高蛍光クローン、(B)中~低蛍光クローン、(C)CHO-K1ネガティブコントロール。
これらの結果は、膜結合Gタンパク質共役ムスカリン受容体の発現レベルと機能活性との間に正の相関があることを示しています。CHO-K1ネガティブコントロール群でカルシウム蛍光信号が検出されなかったことは、ClonePix 2システムがM1 GPCRの細胞表面発現の有無を正確に識別できることをさらに裏付けています。
結論
ハイスループットスクリーニングと選択性プロファイリングの要求に応えるため、GPCRトランスフェクト細胞株は、機能セルベースアッセイにおける独自で堅牢かつ均一なプラットフォームです。今回の予備研究により、ClonePix 2システムがGPCRクローンの発現レベルの違いを確実に検出できることが明らかになりました。さらに、蛍光強度は、膜結合Gタンパク質共役ムスカリン受容体M1の発現差によって引き起こされる細胞質カルシウムレベル変化の大きさと正の相関を示し、FLIPR Tetraシステムで測定されました。
したがって、ClonePix 2システムは、GPCR発現クローンを効率的に検出・ピックし、自然なエピトープを持つ抗原を用いた抗体作製や、構造的に特徴的なGPCRファミリー由来の発現困難なGPCRに対するセルベース機能アッセイなど、さまざまな用途に対応する高品質GPCRタンパク質の独自ソースを提供できます。
ClonePix 2自動動物細胞コロニーピッキングシステムについてさらに詳しく >>
PDF版(英語)