Application Note ヒトiPSCニューロンを用いた3D培養で
神経発達を評価するハイコンテント解析
- 化合物スクリーニングのための3D神経毒性アッセイを開発
- ハイドロゲルとハイコンテントイメージングを活用し、ハイスループットな3D神経突起伸長アッセイを実現
- IC₅₀値の算出や複数化合物の毒性比較に利用できる定量的測定データを生成
PDF版(英語)
はじめに
化合物スクリーニングのために、より複雑で生物学的に関連性が高く、予測性のある細胞ベースアッセイを開発することは創薬における主要な課題です。3Dアッセイモデルの開発と統合は、トランスレーショナルバイオロジーを推進するためにますます普及しています。特に、3D培養はヒト組織の構造、細胞の配置、細胞間および細胞-マトリックス間相互作用、より生理学的に関連する拡散特性などを忠実に再現できるという利点があります。
ハイドロゲルは、神経細胞を3D環境で成長させるために細胞外マトリックスを模倣する目的で広く使用されています。このハイドロゲルはポリエチレングリコール(PEG)をベースとした完全合成で透明です。ハイドロゲルは、MMP切断部位を組み込み、細胞接着をサポートするRGDモチーフを含むことで細胞移動を可能にしています¹。完全合成ハイドロゲルは、96ウェルプレートにあらかじめキャストされ、表面密度勾配を備えており、ハイドロゲル表面に播種された細胞が3Dに浸潤することを促進します(3DProSeed™ハイドロゲル、Ectica Technologies)。このハイドロゲルプラットフォームは使いやすく、自動化にも対応しています *2,3(図1)。
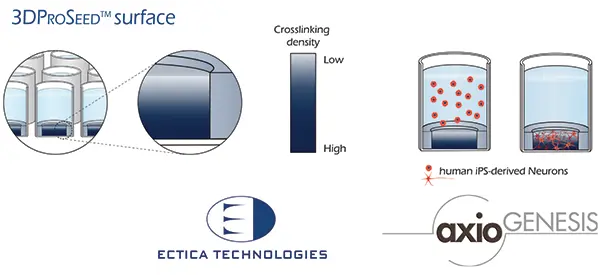
図1. 3DProSeed™ハイドロゲルとニューロン細胞のマトリックス内播種の模式図
さらに、ヒト誘導多能性幹細胞(iPSC)由来ニューロンは、生理学的な細胞モデルの開発にますます利用されています。ヒト由来であること、培養中の長期生存性、無制限の供給可能性により、神経科学分野において初代細胞や動物モデルと比較して優位性があります。
マルチウェルマイクロタイタープレートでの3D神経突起ネットワーク形成
96ウェルフォーマットでの細胞培養と染色を最適化し、3Dマトリックスで成長したニューロンの形態学的フェノタイプと生存率を評価するための共焦点イメージングおよび解析プロトコールを開発しました。
アッセイに使用した細胞はCNS.4U™(Axiogenesis AG)で、ヒトiPSC由来のニューロン(グルタマターギック、ドーパミナーギック、GABA作動性)とアストロサイトの混合物です。CNS.4U™細胞は3DProSeed™ハイドロゲル(Ectica Technologies)で最大14日間培養しました。細胞は1ウェルあたり40,000ニューロンを200 μLの培地で播種しました。選択された研究では、細胞播種密度の影響も評価しました(5,000~80,000 cells/well)。培地組成は、Neurobasal培地とDMEM/F12の50:50混合にサプリメントを加えたものです(Axiogenesis)。
細胞はハイドロゲル表面に播種され、その後ハイドロゲル内部に浸潤しました(図1)。神経突起の伸長は播種後約24時間で開始し、培養14日間にわたり継続しました。神経突起ネットワークの形成は透過光イメージングで経時的にモニタリングしました。エンドポイント測定では、細胞を4%ホルムアルデヒドで固定し、0.1% Triton X-100で透過処理後、TuJ-1ニューロンマーカーに対する蛍光抗体とHoechst核染色で染色しました。
画像スタックから3D画像を生成
ハイコンテントイメージングと解析を用いて、ニューロンネットワーク形成に対する処理効果を評価しました。焦点軸(Z軸)に沿って異なる平面で画像を取得しました(Zスタック)(図2)。画像はImageXpress® Micro Confocal ハイコンテントイメージングシステムを使用し、10Xまたは4X対物レンズで取得しました。Zスタックは5~10 μm間隔で11~33平面を取得し、深さ150~300 μmをカバーしました。2D投影(最大投影またはベストフォーカス)画像は自動的に保存され、すべての個別画像も保存され3D解析に使用されました。
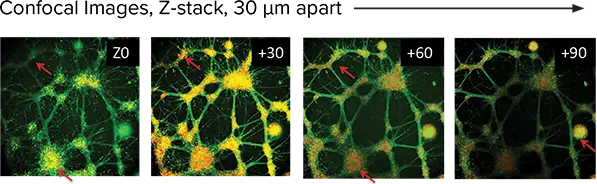
図2. CNS.4U™細胞を3DProSeed™に40,000細胞/ゲルで播種し、14日後に共焦点モードでイメージング 核はHoechst(赤)、チューブリン–TuJ-1(緑)で染色。4つのパネルは異なる焦点面を示し、神経突起ネットワークがハイドロゲル内に数百μm浸潤している様子を示します。
2Dまたは3D画像セットからの測定値生成
画像解析には、MetaXpressソフトウェアに搭載された3D解析モジュールを使用しました。定量解析は2つの方法で実施しました:投影画像(2D)の解析と3D解析です。Hoechst染色およびTuJ-1蛍光を含む2D最大投影画像は、神経突起伸長アルゴリズムを用いて解析しました。フェノタイプの読み出しには、神経ネットワークの広がりと複雑性を定量的に特徴づけるため、神経突起伸長、突起数、分岐数、細胞数および生存率などの多重読み出しを含めました。透過光画像の解析にはベストフォーカス投影画像を使用しました。投影画像の解析により、これらのフェノタイプを迅速かつ正確に定量化できます。図3は、透過光、蛍光(TuJ-1および核染色)、および神経突起伸長測定に使用された解析マスクの画像例を示します。
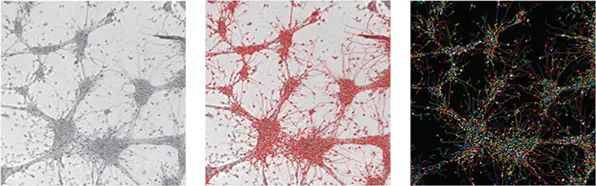
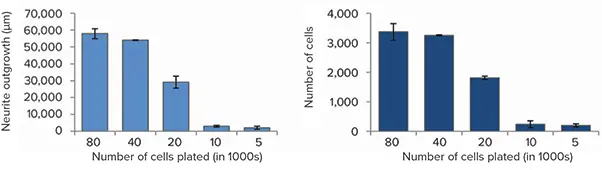
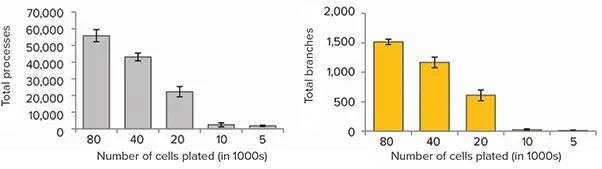
図3. 2D投影画像の解析(上)透過光投影画像とニューロン検出用解析マスクのオーバーレイ。CNS.4U™細胞を異なる播種密度(5,000~80,000 cells/gel)で播種し、14日間培養後に染色・イメージング。Zスタックによる3Dイメージングを実施し、解析には蛍光画像の最大投影と透過光画像のベストフォーカスを使用。自動解析により細胞数、神経突起長、分岐数を測定。グラフは播種細胞数に対する各測定値の依存性を示します(3ウェル平均)。
MetaXpressの3D解析モジュールは、異なる平面のオブジェクトを統合して細胞とネットワークの3D可視化を生成します。3D可視化では、細胞核(擬似カラー)とTuJ-1陽性の神経突起および細胞体(緑)を表示します(図4)。MetaXpressカスタムモジュールエディタを使用して、神経突起と細胞核を定義しました。オブジェクトは各平面で検出され、その後「connect by best match」機能を用いて3D空間で接続されます。3D解析により、マトリックス全体での細胞のより正確な定量化が可能となり、重なり合うオブジェクトも評価できます。
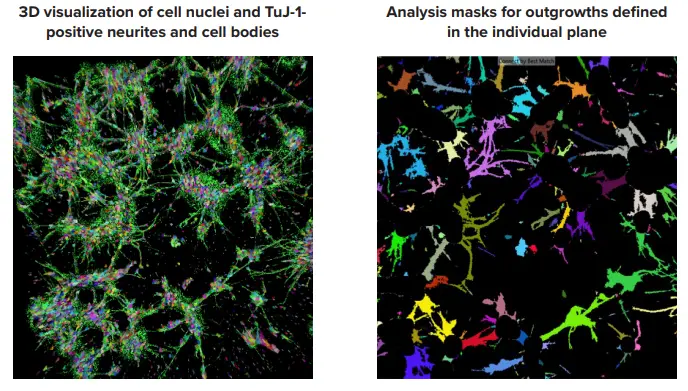
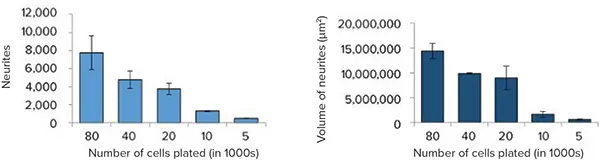
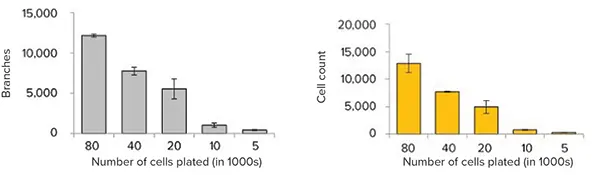
図4. MetaXpressソフトウェアによるハイドロゲル内の細胞とネットワークの3D評価。左上:3D可視化では細胞核(擬似カラー)とTuJ-1陽性の神経突起および細胞体(緑)を表示。右上:個々の平面で定義された突起の解析マスク。3D解析ではウェルあたりの神経突起数、総突起体積、分岐点数、細胞数(核)を定量化。グラフは播種細胞数に対する測定値の依存性を示します(3ウェル平均)。
3Dハイドロゲルを用いた化合物スクリーニング
フェノタイプの読み出しには、神経ネットワークの広がりと複雑性を定量的に特徴づけるための多重読み出しを含めました。アッセイの再現性を評価し、複数の測定値を特徴づけ、既知の神経毒性化合物を用いて試験しました。解析には、標準的な神経突起伸長アルゴリズムを用いた投影画像解析と、神経突起、分岐、核を定義するカスタムモジュールを用いた3D解析の2つの方法を比較しました。
ハイドロゲルでの神経毒性試験の実現可能性を評価するため、播種72時間後に化合物を添加し、処理後7~10日間にわたり効果を測定しました(図5)。培地は2日ごとに交換しました。処理後、培養物を固定、染色し、前述の方法でイメージングしました。選択した化合物の神経ネットワーク(神経突起伸長およびその他の読み出し)に対する濃度応答効果を測定しました。これらの実験は、神経毒性評価に本アッセイシステムを使用するための概念実証を提供します。
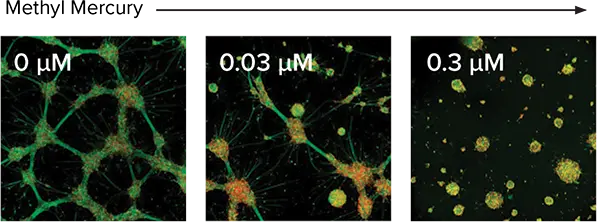
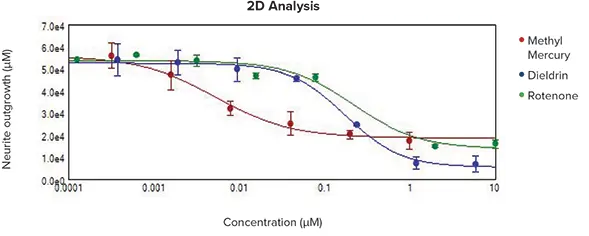
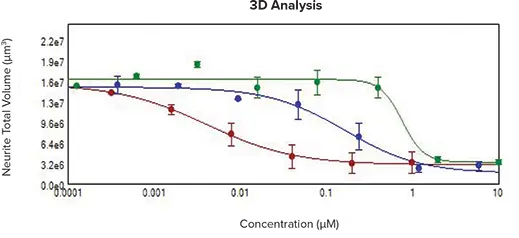
図5. 化合物の濃度応答測定。播種72時間後に既知の神経毒性化合物3種を0~10 μMの濃度で添加。上:神経突起発達に対する阻害効果が視覚的に確認可能。上部グラフ:蛍光画像の最大投影を用いた解析による濃度応答曲線。メチル水銀(IC₅₀ 5 nM)、ディルドリン(IC₅₀ 170 nM)、ロテノン(IC₅₀ 220 nM)。IC₅₀値は3D解析および既報データと同等。下部グラフ:3D解析結果。総突起体積を測定し、分岐総数の濃度応答曲線を表示:メチル水銀(赤、IC₅₀ 2 nM)、ディルドリン(青、IC₅₀ 170 nM)、ロテノン(緑、IC₅₀ 750 nM)。
まとめ
本研究では、3Dニューロン培養における生存率と形態変化を評価可能な定量的ハイスループットアッセイを開発しました。3DProSeed™ハイドロゲル、CNS.4U™ヒトiPSC由来神経細胞、および共焦点ハイコンテントイメージングを用いることで、本手法はハイスループットな化合物毒性スクリーニングおよび安全性評価に利用できます。
高解像度イメージングと多パラメータ解析により、神経突起およびシングルセルのカウントが可能となり、3Dにおける神経突起発達と分岐を統計的に特徴づけます。2Dおよび3D解析を用いることで、IC₅₀値の算出や複数化合物の毒性比較に利用できる定量的測定が可能です。
参考文献
- Ehrbar, M.; Rizzi, S. C.; Hlushchuk, R. et al., Biomaterials 2007, 28 (26), 3856-66.
- Simona B.R. et al., Biomater. Sci., 3:586-591, 2015.
- Zhang N. & Milleret V., SLAS Discovery, accepted, 2017.
- Sirenkoら、Assay and Drug Dev Technologies12(9-10):536-47
ハイコンテントイメージングシステムについて詳しくはこちら>>
PDF版(英語)