Application Note CloneSelect Imager による
ラベルフリーイメージングの多様な応用
- ハイスループットアプリケーションのニーズに応える高速取得機能
- 顕微鏡の専門知識がなくても使える、直感的な操作性のソフトウェア
- コロニー計数、コンフルエンス測定、モノクロナリティ評価・レポート作成のための事前設定済みアルゴリズムを搭載
- オムニトレイから384ウェルマイクロプレートまで、さまざまなプレート形式に対応
PDF版(英語)
はじめに
Sarmad Al-Bassam博士|アプリケーションサイエンティスト|モレキュラーデバイス
近年、対物レンズ、カメラ、ソフトウェアの進歩により、顕微鏡はライフサイエンス分野の創薬や製造現場において、一般的かつ日常的な作業の自動化を可能にしています。しかし、自動化顕微鏡での撮像設定には、時間と労力、そして専門的なトレーニングが必要となる場合が多く、導入のハードルとなっています。また、撮像時間のばらつきは、研究開発チームの生産性やスループットに影響を与える要因にもなり得ます。さらに、下流の解析に適した高品質なデータを取得するためには、露光条件やフォーカス面の最適化といった調整も重要であり、これもまた技術的な課題の一つです。
ラベルフリーでマイクロプレート上の生物学的情報を定量化することには、いくつかの技術的な課題があります。まず、ラベルフリー画像はシグナルと背景のコントラストが低いため、解析が困難になることがあります。また、広い視野を必要とする生物学的対象を解析する場合、多数のタイル画像を取得する必要があり、これも一般的な課題の一つです。さらに、複数のタイムポイントで断続的に取得されたウェル画像とメタデータをリンクさせる作業も、解析の複雑さを増す要因となります。
CloneSelect™ Imager は、透過白色光を用いたさまざまなアプリケーションに対応可能な自動撮像装置です。操作が簡単で、顕微鏡の使用経験がない複数人の研究チームでも安心してご利用いただけます。また、高速な画像取得により、大規模なグループでのハイスループットニーズにも対応します。本装置は独自の真空ベース設計を採用しており、さまざまなプレートタイプにおいて常にピントの合った画像を取得できます。1枚のプレートを約90秒で撮像できるため、効率的なデータ収集が可能です。さらに、ウェル全体を対象とした解析や、複数のタイムポイントにわたる生物学的な課題にも適しており、柔軟な研究設計をサポートします。本ページでは、CloneSelect Imager を活用した代表的なラベルフリーイメージングの応用例と、各種マイクロプレート形式における有用性についてご紹介します。
結果
6ウェルプレートでのコロニーカウント
ラベルフリーイメージングの代表的な応用の一つに、マイクロプレートのウェル全体におけるオブジェクト(例:コロニー)の計数があります。典型的な例としては、**クローン原性アッセイ(colony formation assay)**が挙げられます。これは、半固形培地中で単一細胞がコロニーへと成長する能力に基づいた、in vitro における細胞生存率評価法です。また、スクリーニング用途として、半固形培地中でのコロニー成長速度の評価も一般的な応用例です。こうしたスクリーニングでは、分泌能や結合能の高いターゲットを不均一な細胞集団から選別することが目的となります。これらのアプリケーションでは、6ウェルプレートがよく使用されます。これは、スループットとウェル面積のバランスが取れているためです。このような用途では、プレートのウェル全体を迅速に撮像し、得られたデータに対して客観的な解析を行うことが極めて重要です。解析には、コロニー数やサイズの定量化が含まれ、生データおよびメタデータのエクスポートによって、さらなる解析にも対応可能です。
CloneSelect Imager は、6ウェルプレートを用いたコロニーの可視化および特性評価に対応しています。一例として、SJK-285 マウスハイブリドーマ細胞を含む 6ウェルプレートを 7日目にスキャンした画像が図1に示されています。プレート内の各ウェルの内容は、Plate Thumbnails タブに表示され(図1B)、このビューではコロニーの存在を検出するアルゴリズムが緑色で示され、背景は黒色で描画されます。各サムネイルをクリックすると、ウェル全体の画像ビューに切り替わります。このビューでは、4倍対物レンズで撮影された画像が 10×10 のマトリックス状にタイル配置され、ソフトウェアによって1つのウェルとして解析されます(図1C)。
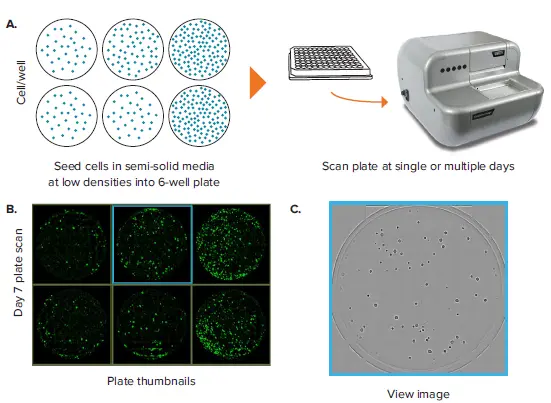
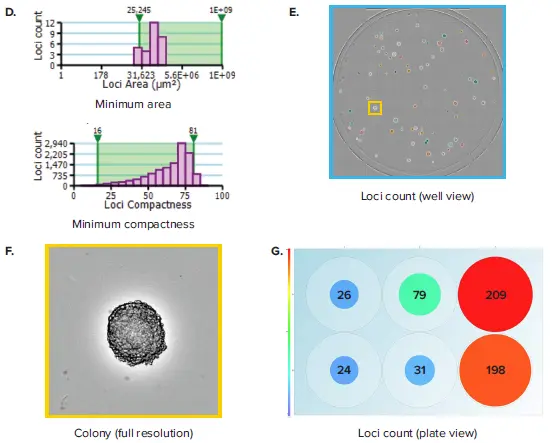
図1. 6ウェルプレートにおけるコロニー計数 (A) ハイブリドーマ細胞をそれぞれ 50、100、200細胞/ウェルで播種し、7日間培養した 6ウェルプレートを撮像。(B) Plate Thumbnailビューでは、プレート全体が表示され、コロニーは黒背景上に緑色の疑似カラーで識別されます。(C) View Imageウィンドウでは、ウェル全体の透過白色光画像が表示されます。(D) コロニーの面積とコンパクトさ(円形度の指標)に基づくゲーティング範囲を調整することで、セグメンテーションおよびコロニー計数の条件を変更可能です。(E) 面積とコンパクトさの最適なゲーティングに基づく、ウェル全体でのコロニーセグメンテーション。(F) ウェルビュー内をズームインすることで得られる、コロニーの高解像度画像。(G) プレートビューでは、サイズに応じた色付き円でコロニー数を表示。赤色の大きな円はコロニー数が多いウェルを、青色の小さな円はコロニー数が少ないウェルを示します。
CloneSelect Imager は、約90秒でプレート全体をスキャンできる高速撮像機能に加え、リアルタイム解析機能により、効率的かつ客観的な画像の定量評価を可能にします。Loci Count タブに移動すると、ウェル内のオブジェクトを**サイズ(面積)およびコンパクトさ(円形度)**に基づいてゲーティングすることができます(図1D)。ユーザーはこれら2つのパラメータのゲート閾値を調整することで、すべてのウェルにおいて適切にコロニーをセグメントできる条件を設定できます(図1E)。この設定は、複数のウェルを切り替えながらセグメンテーションの有効性を確認することで検証できます。最終的に、Loci Count タブ内のプレートビューに戻ることで、各ウェルのコロニー数を視覚的に確認できます。また、データエクスポートボタンを使用することで、各プレートに関する関連データをエクスポート可能です。エクスポート形式は CSV、XML、Excel など複数に対応しています。表1には、各ウェルの**コロニー数(loci count)および平均コロニー面積(mean loci area)**を含む、代表的なエクスポート例が示されています。さらに、異なるタイムポイントで複数回スキャンを行うことで、日ごとのコロニーサイズの変化を追跡することができ、これらのデータを集約することで、各タイムポイントにおける平均コロニーサイズの評価が可能となります。
| ウェル | 遺伝子座数 |
平均遺伝子座面積 (μm²) |
|---|---|---|
| A1 | 26 | 161813 |
| A2 | 79 | 217003 |
| A3 | 209 | 147454 |
| B1 | 24 | 198058 |
| B2 | 31 | 265671 |
| B3 | 198 | 146133 |
表1. コロニー(遺伝子座)数とウェルあたりの平均遺伝子座面積を示す、図1のプレートからエクスポートしたデータ。
96ウェルおよび384ウェルプレートでのモノクローナリティ評価
コロニー計数に加えて、ラベルフリーイメージングの一般的な応用として、モノクロナリティ評価およびクローン増殖の特性解析があります。 このアプリケーションでは、対象となる細胞集団に対してリミティング・ディリューション(希釈播種)を行い、プレート内の一部のウェルのみにシングルセルが播種されるように設定します。この用途では、96ウェルプレートや384ウェルプレートが一般的に使用されます。これは、ハイスループットのニーズと、1536ウェルプレートにおけるシングルセル播種効率の制限とのバランスが取れているためです。リミティング・ディリューションでは、細胞がポアソン分布に従って播種されるため、後にシングルセル由来のクローンを分離することが可能になります。
CloneSelect Imager は、モノクロナリティ関連のアプリケーションにおいて、迅速なデータ取得を可能にします。96ウェルプレートや384ウェルプレートのスキャンは、標準解像度(\( \frac{3.7\ \mu\text{m}}{\text{pixel}} \))で約90秒という短時間で完了します。撮像後は、Plate Thumbnails タブにアクセスすることで、プレート全体のビューから各ウェルへと簡単にナビゲートできます(図2B、2D)。このインターフェースでは、単一コロニーを含むウェルをクリックして選択することができます(図2C、2E)。指定されたコロニーがシングルセル由来かどうかを確認したい場合は、Report Generator タブにアクセスします。このタブでは、複数の日に取得されたスキャン画像がリンクされており、単一コロニーが確認されたタイムポイントから、初期のシングルセル播種時点まで遡ってウェルの状態を確認することができます。各タイムポイントにおいて、Cell Region(黄色の枠、図2C、2E)を指定・調整することで、指定ウェルに対するモノクロナリティレポートの自動生成が可能になります。図2Fには、複数のタイムポイントにおける Cell Region の例が示されており、これらはエクスポートデータにも一般的に含まれます。
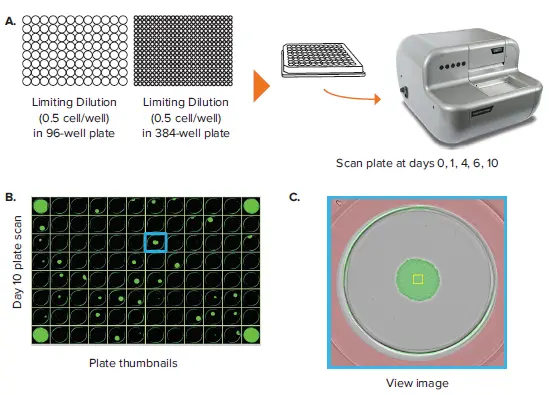

図2. 96ウェルおよび384ウェルプレートにおけるモノクロナリティ評価 (A) 細胞は、\( \frac{0.5\ \text{cell}}{\text{well}} \)の密度で96ウェルまたは384ウェルプレートに播種されます。プレートは、Day 0から開始して複数のタイムポイントでCloneSelect Imagerによってスキャンされます。(B) リミティング・ディリューションから10日後の 96ウェルプレートの Plate Thumbnails ビュー。(C) 96ウェルプレートのウェル全体画像。黄色の枠は Cell Region を示します。(D) リミティング・ディリューションから10日後の 384ウェルプレートの Plate Thumbnails ビュー。(E) 384ウェルプレートのウェル全体画像。(F) Report Generator は、ユーザーが定義した Cell Region に基づいて、Day 0 から Day 10 までの細胞画像を時系列で表示するフィルムストリップを自動生成します(パネルCの細胞画像を含む)。
96ウェル・ハーフエリアプレートにおけるコンフルエンスの測定
もう一つの重要なラベルフリーアプリケーションとして、ウェル内のコンフルエンス(細胞の密度)の評価があります。コンフルエンスはエンドポイントで評価されることがあり、その場合は実験終了時に静的なタイムポイントでスキャンを行います。一般的には96ウェルプレートが使用されますが、近年ではスループットの向上や培地・試薬のコスト削減のために、96ウェル・ハーフエリアプレートを選択する研究者が増えています。ある実験の例では、96ウェル・ハーフエリアプレートを用いて細胞集団の段階希釈を設定します。最上段のA列には\( \frac{10{,}000\ \text{cells}}{\text{well}} \)から開始し、B列からH列にかけて2倍ずつ希釈していきます(図3A)。図3Bは「Plate Thumbnails」タブの表示を示しています。ソフトウェアでは、細胞種や形態に応じて3種類のしきい値アルゴリズムから選択することが可能です。適切なしきい値アルゴリズムを適用することで、コンフルエンス率をリアルタイムで計算できます。「Confluence」タブに移動すると、プレート全体のコンフルエンスデータを円グラフ形式で視覚化できます。円グラフが完全に塗りつぶされている場合、そのウェルのコンフルエンスは100%であることを示します(図3C)。このコンフルエンスデータから、ウェルごとの細胞数を推定することも可能です(図3D)。細胞数の推定には、特定の細胞種に対して既知の播種密度に基づいた標準曲線の作成が必要です。
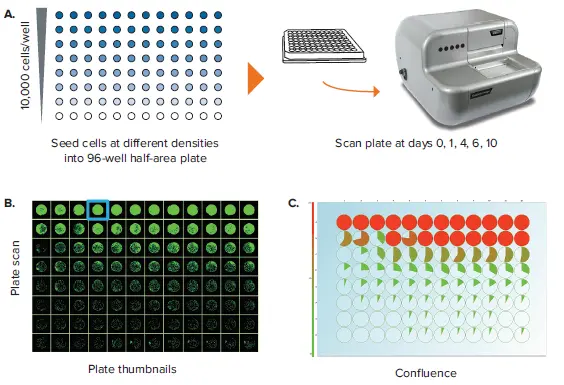
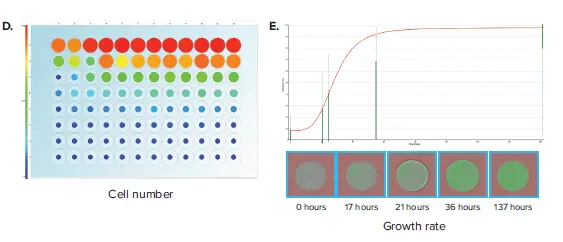
図3.96ウェル・ハーフエリアプレートにおけるコンフルエンス評価 (A) 細胞は、96ウェル・ハーフエリアプレートに\( \frac{10{,}000\ \text{cells}}{\text{well}} \)から開始して、密度を段階的に減らしながら播種されます。プレートは、初回播種時から複数のタイムポイントでCloneSelect Imagerによりスキャンされます。(B) 「Plate Thumbnails」ビューでは、コンフルエンスのパターンが表示されます。緑色の疑似カラーは細胞の存在を示し、黒色はバックグラウンドを示します。(C) 「Confluence」タブビューでは、コンフルエンスの定量結果が円グラフ形式で表示されます。(D) 「Cell Number」タブでは、既定の標準曲線に基づいて推定された細胞数の視覚化が表示されます。(E) 「Growth Rate」ビューでは、ウェルごとのコンフルエンス率が時間に対してプロットされます。赤い曲線は全ウェルの平均値を示します。また、ウェル画像のフィルムストリップには、異なるタイムポイントでのウェルのコンフルエンス画像が表示されます。
または、コンフルエンスは一般的に動的なプロセスとして適用されます。この場合、複数のタイムポイントでスキャンを取得し、データをプロットして成長曲線を生成します。このアプリケーションでは、コンフルエンスは細胞集団の増殖特性を評価する上で重要であり、細胞株の開発において重要な役割を果たします。例としては、低増殖クローンを除外するために、リミティング・ディリューションまたはシングルセルプリンティングによって得られたシングルセルウェルからの増殖特性の評価や、コロニーピッキング後の細胞集団の増殖速度の評価などが挙げられます。「Growth Rate」タブに移動することで、さまざまな取得タイムポイントをリンクすることができます(図3E)。研究者は、特定のウェルの増殖速度がプレート内の他のウェルとどのように比較されるかを確認できます。コンフルエンスおよび細胞数のデータは、複数のタイムポイントにわたって共通フォーマットでエクスポートし、さらなる解析に使用することが可能です。
結論
ラベルフリーイメージングは、マイクロプレート内の生物試料を評価するための、最も直接的かつ非侵襲的な方法です。一部のアッセイでは、ラベルフリーイメージングによってマイクロプレート内の細胞増殖を非侵襲的に観察することができ、研究者は実験を論理的なエンドポイントまで進めることが可能になります。これに対して、比色法や蛍光イメージングアッセイでは、マーカーの固定や染色が必要となるため、観察時点で実験が終了してしまいます。ラベルフリーイメージングは、細胞株開発プログラムなどのバイオ医薬品製造環境において、特に有用なアプリケーションの一つです。
CloneSelect Imagerは、複数の検証済みプレートフォーマットにおいて、固定された焦点高さで迅速なラベルフリーイメージングを行うのに理想的なシステムです。本システムでは、6ウェルプレート、96ウェル標準プレートおよびハーフエリアプレート、384ウェルプレートなど、多様なプレートフォーマットを用いたさまざまなアプリケーションが実証されています。さらに、オムニトレイ、ラウンドボトムプレート、12ウェル、24ウェル、48ウェルプレートなど、追加のプレートフォーマットにも対応可能です。本システムは、1枚のプレート全体を約90秒でイメージングすることができ、オンザフライの画像解析によって、画像ベースのデータから客観的なインサイトを得ることができます。細胞およびオブジェクトのセグメンテーションに関する既定のアルゴリズムにより、生物学的アプリケーションにおいて精密かつ正確な測定が可能です。ラベルフリーデータの客観的解析により、今後の実験に向けて一貫性のある、情報に基づいた意思決定を行うことができます。
PDF版(英語)