Application Note MetaXpress透過光解析モジュールによるセルカウント
- 透過光画像による対物レンズのセグメンテーションとカウント
- 透過光による細胞増殖のモニタリング
- TL Cell Segmentation Algorithmとマルチアッセイの効果を評価
- 複数の細胞タイプとサイズの定量化と解析のためのツールを理解
PDF版(英語)
はじめに
Matthew Hammer|アプリケーションサイエンティスト|モレキュラーデバイス
標識不要の細胞アッセイは、細胞数、増殖、健康状態、コンフルエンス、細胞毒性をモニターする多くの生物製剤に必要とされています。このようなアプリケーションでは、効率的でロバストな透過光(TL)イメージングと解析機能が必要であり、細胞を正確にセグメンテーションして定量し、さまざまな細胞の反応や形態を評価する必要があります。さらに、高コントラスト透過光イメージングと蛍光標識およびイメージングの結合は、多くの細胞ベースのアッセイに不可欠です。
MetaXpress® 6.5カスタムモジュールエディター内のTL Cell Segmentation Algorithmは、透過光を用いてイメージングされた細胞の定量化のための最適化可能で信頼性の高い解析アルゴリズムです。このアプリケーションノートでは、透過光の全細胞数と蛍光染色した核数を比較することにより、CHO細胞およびHeLa細胞の解析と定量におけるTL Cell Segmentation Algorithmの有用性と精度を実証します。図1
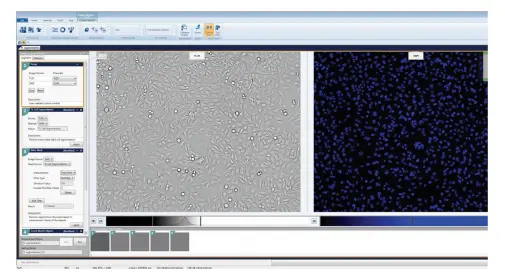
図1. 透過光で捉えたHeLaとCHO細胞を分析するためのカスタムモジュールのセットアップです。細胞はHoechst 33342核色素で染色しました。細胞はTLとDAPIフィルターでイメージングし、透過光イメージングと核の蛍光イメージングに基づいて細胞を解析・定量するようにカスタムモジュールを設定しました。
方法
CHOとHeLa細胞の希釈液を96ウェル、黒色、透明底のマイクロプレートにプレーティングしました。各細胞タイプの希釈液は6連で行い、細胞は1:2でプレーティングしました。プレーティングから24時間後、細胞を37℃、5%CO2インキュベーター内で30分間、Hoechst 33342核色素で染色しました。その後、細胞を4%パラホルムアルデヒドで固定し、DPBSで2回洗浄しました。細胞をImageXpress® MicroハイコンテントイメージングシステムとMetaXpress®ソフトウェアで画像化しました。1視野を10x Plan Fluor対物レンズで撮像し、最大1500個の細胞を正確に捉えました。画像は透過光とDAPI蛍光チャンネルで撮影され、露光時間はそれぞれ15ミリ秒と35ミリ秒でした。
Custom module editorでTL Cell Segmentation Algorithmを設定
透過光で撮影した細胞の定量化には、正確なセルセグメンテーションが不可欠です。このアルゴリズムが最も正確にセグメンテーションし、HeLa細胞とCHO細胞の両方をマスクするため、TL Cell Segmentation AlgorithmにはCell Aが選択されました(図2)。Cell BやCell Cという他の設定もあり、それぞれ小さな細胞や大きな細胞の分析に使用できます。総面積が150 µM2以上でない細胞をフィルタリングするために、モジュールにフィルタリング・マスクを追加しました(図3)。これにより、小さく丸められた死細胞が生細胞としてカウント・分析されないようにしました。さらに、Nuclei Count Objects Algorithmは、HeLa細胞とCHO細胞の核を正確に定量化するように設定されました(図4)。
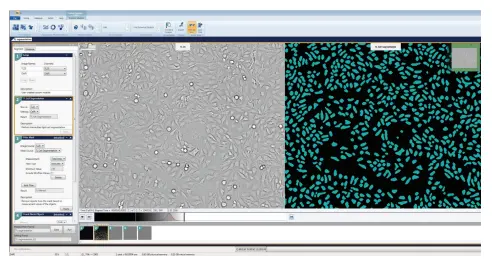
図2. 選択したHeLa細胞の透過光画像にTL Cell Segmentation Algorithmを適用します。Custom Module Editorで、TL Cell Segmentation Algorithmを設定するには、光源波長と細胞タイプを表す設定を選択します。セルAは、撮像されたHeLa細胞とCHO細胞を最適にマスキングしました。
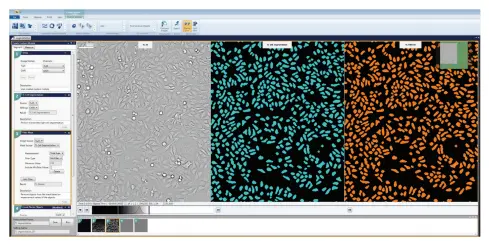
図3. TL Cell Segmentation overlayとFilter Mask overlayの比較画像。フィルタリング・マスクは、150 µm2と設定された最小総面積要件に基づいて細胞をフィルタリングするよう設定されました。
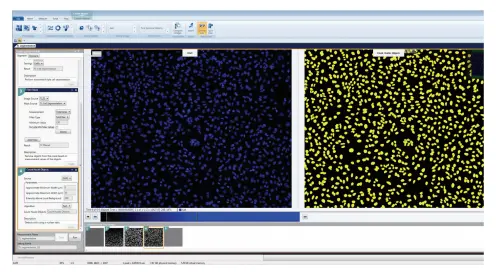
図4. 蛍光核イメージャーのCount Nuclei Objectsセグメンテーション。Count Nuclei Objectsは、Hoechst 33342で染色された核に基づいて細胞を定量化するために利用されました。すべての核を正確にマスクするために、おおよその最小幅と最大幅、およびローカルバックグラウンド以上の強度のパラメータが最適化されました。(左)核は青色、(右)解析マスクは黄色。
透過光での細胞数と蛍光核数との相関性
TL Cell Segmentation Algorithmで構成されたThe Custom Moduleは、HeLa細胞とCHO細胞の両方を正確にセグメンテーションしました(図6)。透過光の総細胞数と核数に統計的な差はなく、TL Cell Segmentation Algorithmの正確さが際立ちました(図7)。細胞数に加え、TL Cell Segmentation Algorithm(図5)により、細胞反応と形態の異なる特性を評価するための複数の読み出し値の生成が可能になりました。例えば、総カウント数、平均カウント数、面積、 強度、積分強度、形状、高さ、幅に加え、細胞や細胞内の対物レンズ など、様々な追加読み出しを生成することができます。真円性死細胞を除外するフィルタリング・マスク(Filter Mask)の使用は、生細胞染色または全細胞染色を使用する必要性を軽減するだけでなく、これをさらに容易にします。さらに、核染色とCount Nuclei Objects Algorithmの使用により、透過光でイメージングされた細胞内の蛍光標識または染色された物体の分析から、複数の読み出しが可能であることが実証されました。
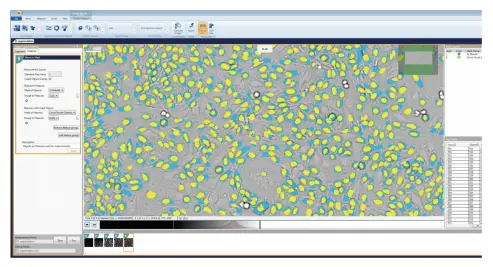
図5. Custom Moduleで設定した画像解析マスクを重ねたHeLa細胞の代表画像。青色マスクはTL Filter Maskを用いたTL Cell Segmentation Algorithmで検出された細胞を表し、黄色はCount Nuclei Objects Algorithmで検出された核を表します。Custom Module で必要な測定は、Measure タブで設定しました。対物レンズは TL フィルタリングされたCell Segmentationでマスクされたものとして定義され、 Count Nuclei Object maskは各対物内の Feature を測定するために選択されました。
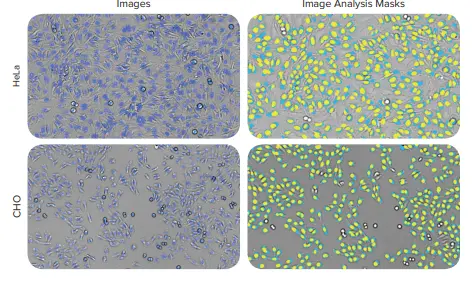
図6. HeLa細胞とCHO細胞のイメージングと、最適化された TL Cell Segmentation Custom Moduleからの画像解析マスクです(左)。DAPI核は青色、(右)透過光中の細胞は水色でマスクされています。画像はImageXpress Microシステムで10x Plan Fluor対物レンズで撮影され、新しいTL Cell Segmentation algorithmで構成されたCustom Moduleで解析されました。
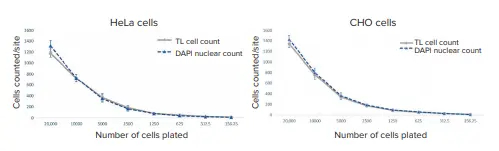
図7. TL Cell Segmentation and Count Nuclei Objectsモジュールに基づくHeLa細胞とCHO細胞の総細胞数の比較です。細胞は1:2に希釈され、ウェルあたり20,000細胞の濃度からスタートしました。
結論
MetaXpressの Custom Module EditorのTL Cell Segmentation Algorithmは、複数の細胞タイプとサイズの定量と解析のための効果的なツールを提供します。ImageXpress®Micro Confocalハイコンテントイメージングシステムによる最適なセグメンテーション機能により、蛍光色素に依存しない形態の解析が可能となります。透過光画像を用いて細胞を定量化する機能は、研究者のアッセイ能力に柔軟性を与えます。ここで説明する方法は、TL Cell Segmentation Algorithmが、増殖、細胞毒性研究、トランスフェクションを含むがこれらに限定されない複数のアッセイに使用できることを実証しています。
PDF版(英語)