Application Note Organ-on-a-chipの自動化:
血管新生のイメージング解析
- アッセイのスループットを向上させます
- データの再現性を改善します
PDF版(英語)
Angeline Lim, Oksana Sirenko | モレキュラーデバイス
Arthur Stok, Matthew Delport | Mimetas
Francis Enane | Beckman Coulter Life Sciences
はじめに
3D細胞モデルは、2D単層で培養された細胞よりも細胞の3Dマイクロ環境をより忠実に再現できるため、多くの研究分野でますます人気が高まっています。現在、化合物が臨床試験で高い失敗率を示す理由の一つは、2D細胞培養システムの不十分さにある可能性があります。そのため、創薬においてより生理学的に関連性の高いモデルを用いて、より良い薬剤候補の選択を可能にすることが求められています。
細胞生物学における新しい手法とバイオエンジニアリングの革新により、3D培養の利用が拡大しています。これらの3D細胞モデルには、単純なスフェロイドから、オルガノイドやorgan-on-a-chip(OoC)システムといったより複雑な構造まで含まれます。3Dモデルの複雑さが増すことは、研究や創薬スクリーニングにおける広範な採用の障害となっています。具体的には、均一な3D構造の生成、再現性のある細胞播種、そして最終的には3D培養をハイスループットアプリケーションに適応させることが課題です。細胞培養プロセス、エンドポイントアッセイ、および解析の自動化は、3D細胞モデルの利用を促進し、スケールアップするために必要なツールを提供します。
これらの課題に対応するため、私たちはオルガンオンチップモデルに対する自動化手法を開発しました。この自動化手法は、細胞培養の自動化とモニタリングに対応した複数の装置で構成される統合ワークセルを利用します。概念実証として、MIMETAS社のOrganoPlate® 3-lane 64を用いて血管新生モデルを構築しました。このOrganoPlateタイプは、384ウェルのイメージング品質プレートフォーマットに64のマイクロフルイディックユニットを備えており、自動液体ハンドリングやハイコンテントイメージングに非常に適しています。細胞外マトリックス(ECM)の分注と内皮細胞の播種は、液体ハンドラーで実施しました。Wave機能を搭載した自動化対応LiCONiCインキュベーターは、マイクロフルイディックチャネルの連続灌流を可能にします。長期培養では、培地交換などのプロセスを自動化ソフトウェアでスケジュールできます。エンドポイントアッセイの読み出しには、ハイコンテントイメージングと解析を用いて、3Dにおける血管新生スプラウトの定量化を行いました。ここで開発した細胞播種、培地交換、および3D血管構造の成長の定量モニタリングの自動化手法は、化合物スクリーニングや毒性試験など、他のアプリケーションにもさらに適用できます。
方法
OrganoPlate 3-laneにおける3D血管新生モデル
血管新生モデルの構築には、OrganoPlateと呼ばれるハイスループットマイクロフルイディックプラットフォームを使用しました。OrganoPlate 3-laneは、64または40の組織培養チップで構成され、それぞれが3つのチャネルを備えています(図1)。各チップの中央チャネルにはコラーゲンI細胞外マトリックス(ECM)ゲルを播種し、フェーズガイドによってパターン化しました。内皮細胞(初代細胞、細胞株、またはiPSC由来)は各チップの右チャネルで培養され、LiCONiC WaveインキュベーターにOrganoPlateを設置することで、重力による培地の流れをシミュレーションする灌流下で内皮血管を形成しました。左チャネルに血管新生因子のカクテルを添加することで、親血管からの血管新生スプラウトの指向性形成を誘導しました。血管新生スプラウトは1~5日間形成させ、その後固定および染色して定量比較を行いました。血管細胞とスプラウトは4%ホルムアルデヒドで固定し、VE-cadherinに対する一次抗体で染色した後、Alexa488二次抗体(緑)で染色し、核はHoechst(青)で染色しました。
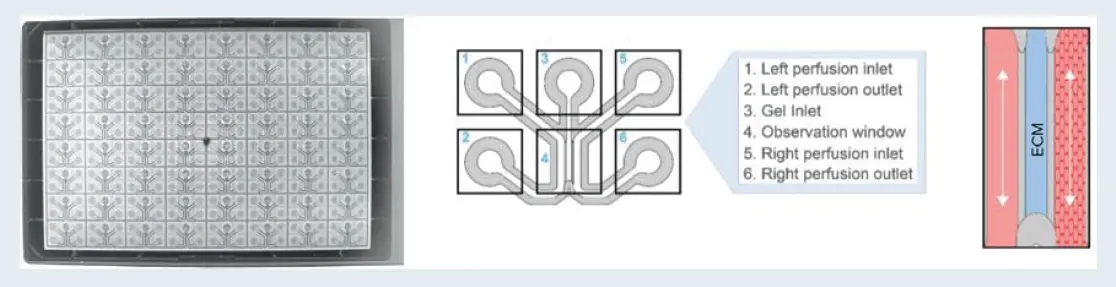
図1. OrganoPlate 3-laneは64または40の培養チップで構成されています。左:OrganoPlate 3-lane 64の底面からのビュー。中央:OrganoPlate 3-lane 64組織チップの模式図。右:ECMゲルに対して成長した細胞チューブのイラスト。

ワークセルの自動化セットアップ
ワークセルは以下の主要コンポーネントで構成: マイクロタイタープレート保管用ホテル、波動機能付きLiCONiC STX44自動インキュベーター、AquaMaxマイクロプレートウォッシャー(モレキュラーデバイス)、HiGTM4自動遠心機(BioNex Solutions)、SpectraMax iD5マルチモードマイクロプレートリーダー、ImageXpress Confocal HT.aiシステム、ImageXpress Pico自動細胞イメージングシステム(モレキュラーデバイス)、Biomek i7自動ワークステーション(Beckman Coulter Life Sciences)。プレートハンドリングロボットはPreciseFlex400ロボットで、すべてのプレートネストにアクセスできるように2mレールに設置されています。Green Button Goスケジューリングソフトウェアは、自動化ワークフローとスケジュール設定の作成に使用されます。このソフトウエアは、ワークセル内の全デバイスを制御するためのシングルユーザーインターフェースをご提供。
OrganoPlateの自動化には、Biomek i7自動ワークステーションを使用してプレート準備(ECM播種)と細胞播種を行いました。Biomek i7ワークステーションは、成長因子の添加、培地交換、細胞の固定および染色にも使用できます。OrganoPlateの搬送は、PreciseFlex400ロボットを使用してBiomek i7液体ハンドラーに移動し、培地交換を行います。インキュベーターからイメージングへの搬送も設定でき、細胞の長期的な間欠モニタリングが可能です。
結果
細胞培養プロセスは通常、複数のポイントでユーザーの介入を必要とする手動手順です。これらのプロセスは、統合ワークセルで自動化できます(図2)。たとえば、マイクロプレートは培地交換のためにインキュベーターから無菌液体ハンドラーに移送できます。さらに、化合物処理やエンドポイントアッセイも同様に自動化できます。細胞培養の維持(モニタリングと給餌)およびエンドポイントアッセイのプロセスを自動化することで、再現性のある結果が得られ、リソースを節約し、高いスケーラビリティを実現します。
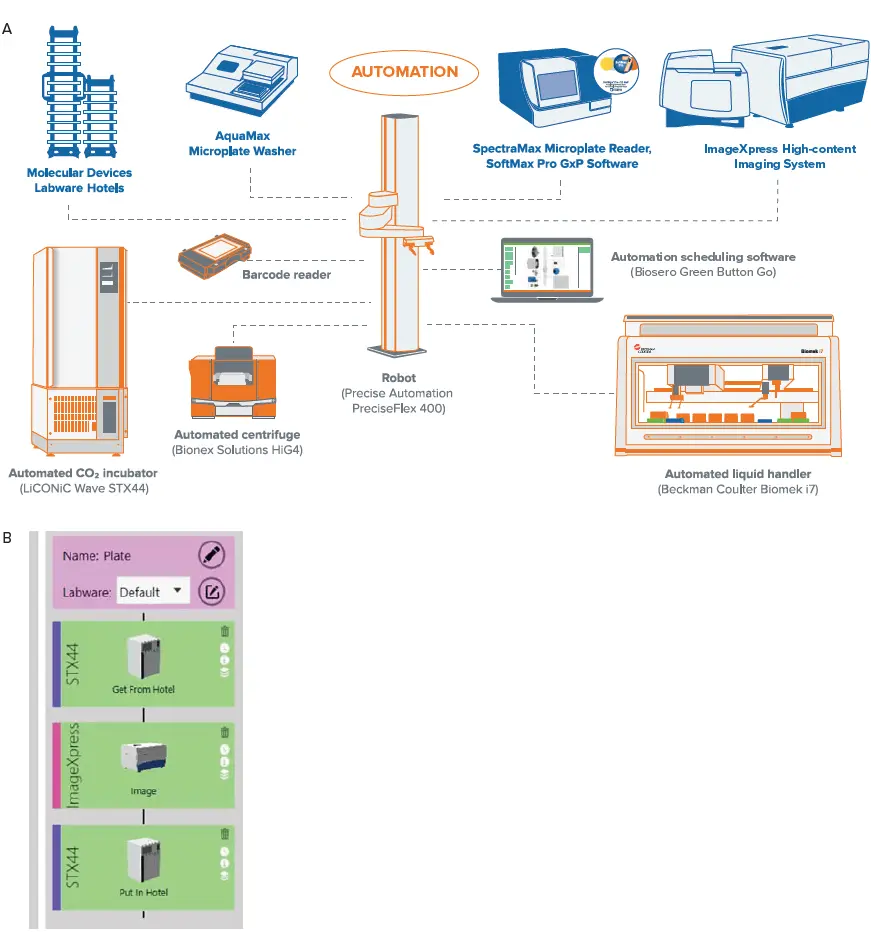
図2. ワークセル内の個々の装置のレイアウトを(A)に示します。装置は統合ソフトウェア(Green Button Go)によって制御され、プロセスの設定が可能です。培養中の細胞をモニタリングするプロセスの例を(B)に示します。ここでは、プレートをインキュベーターからImageXpress Confocal HT.aiに移動し、明視野でイメージングした後、再びインキュベーターに戻します。このプロセスはスケジュール設定が可能で、イメージングが必要なプレートをリストとして入力することで、バッチ処理が容易になります。培地交換(給餌)のための液体ハンドラーを含むより複雑なルーチンも実装できます。
スケーラブルなマイクロフルイディックプラットフォームを用いた血管新生モデルの自動化
OoC技術は、生理学的な臓器とその環境を模倣するために成功裏に使用されています。OoCモデルは、組織の微小環境に関連する重要な生理学的条件、例えば組織間相互作用、細胞パターン化、組織境界、濃度勾配などの制御を可能にします。
ここでは、OrganoPlate 3-lane 64(MIMETAS)を用いて、自動化プラットフォーム上で血管新生スプラウトをin vitroでモデル化しました(図3)。OrganoPlateは灌流と濃度勾配を統合しており、血管新生スプラウトや微小血管の安定化の研究に使用できます。自動液体ハンドリングはアッセイの再現性を改善し、スループットを向上させます。
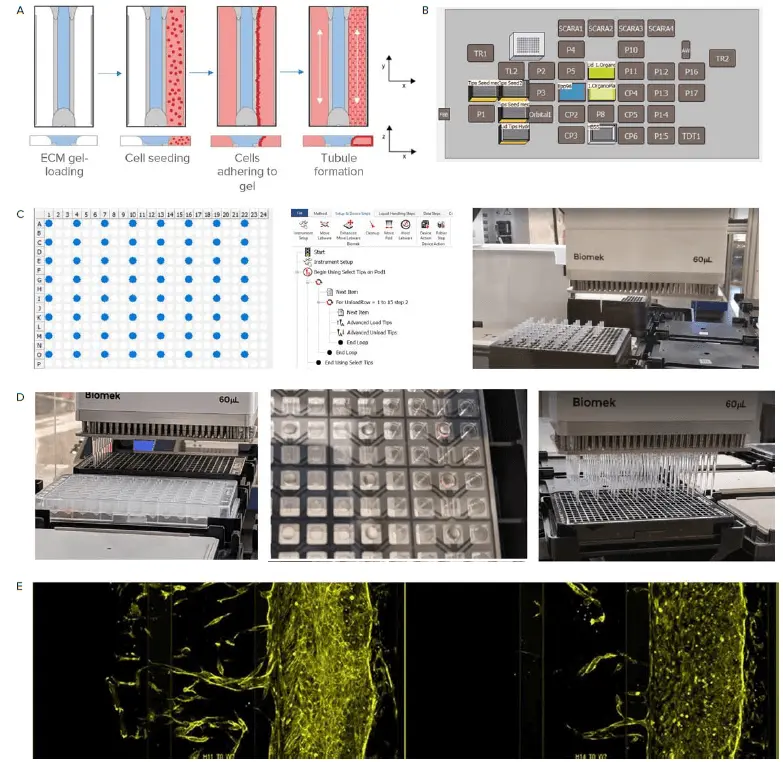
図3. 自動液体ハンドラーによるサンプル準備。A) OrganoPlate準備の手順。観察ウィンドウを示します。青はゲルインレットからロードされるECMを表します。次に、ECMチャネルに隣接する灌流チャネルに細胞を播種します。OrganoPlateは細胞がゲルに沿って沈降し、付着できる角度で配置されます。最後に、プレートをロッカープラットフォーム(またはWave機能付きLiCONiCインキュベーター)に置き、培地灌流を開始し、チューブ形成を可能にします。B) Biomek i7液体ハンドラーは6つのヒーター・クーラーブロック(CP)を備えています。ECMはこれらのCP位置で冷却されます。C) Biomek i7液体ハンドラーは、OrganoPlateフォーマットに合わせてピペットチップを配置できます。D) 左の画像はECMロードを示します。中央の画像は自動化によるゲルロード後のゲルインレットとゲルチャネル内のECMを示します。右の画像は灌流インレットへの細胞培地の添加を示します。E) OrganoPlate 3-lane 64における血管新生スプラウトの経時的形成。
血管新生スプラウトの経時的形成
OrganoPlate 3-laneを用いて血管新生をモデル化しました。内皮細胞はチューブを形成するように播種しました。成長因子はECMを挟んだ反対側のチャネルに添加し、濃度勾配を形成しました。血管新生スプラウトは、成長因子を含むチャネル(下側)に向かって成長する様子が観察されました(図4)。
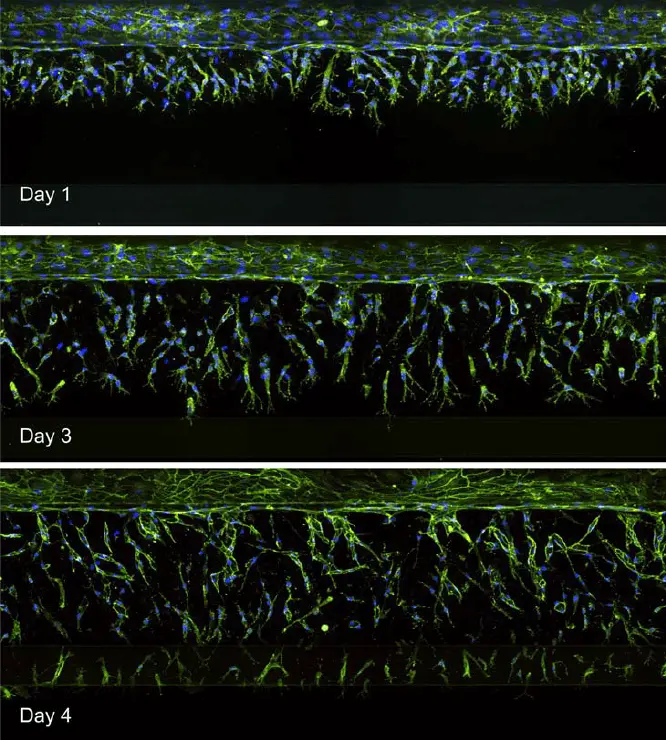
図4. 内皮細胞(上側チャネル)が下側チャネルに向かって成長するイメージング。最大投影画像は血管新生スプラウトの経時的変化を示します。
血管新生スプラウトの3D定量解析
血管新生の時間依存性は5日間にわたりモデル化されました。スプラウトの数と体積の時間依存的な増加、および細胞や核の数の増加が観察されました。画像は3Dで解析され、血管新生スプラウトの成長は、スプラウトと核の総数、強度、体積、オブジェクト間距離など複数の読み出しで特徴付けられました。個々のスプラウトあたりの核の数(または平均)も決定できます(図5)。二次解析はMicrosoft Excelを使用して行いました。
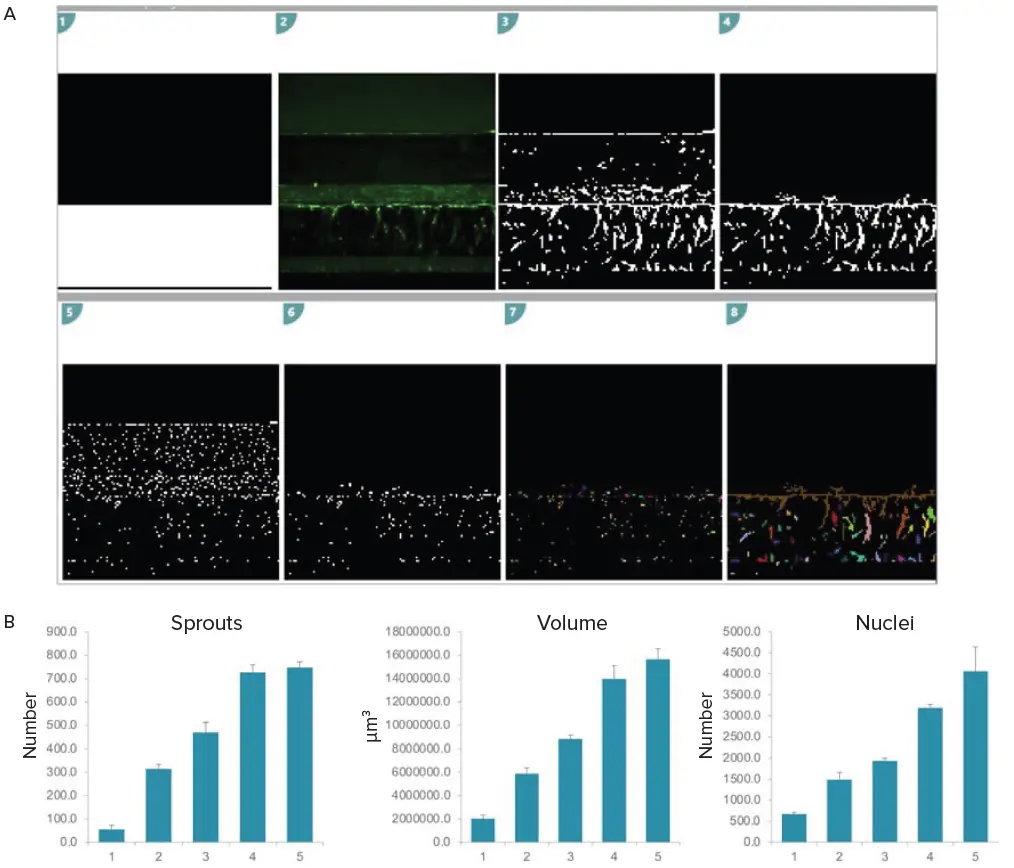
図5. 血管新生スプラウトの3D解析。A) MetaXpress Custom Module Editor(CME)での解析設定。簡単に言うと、ECMチャネルを表す解析領域(白いオーバーレイ)を作成しました。スプラウトのセグメンテーションを改善するために画像の前処理を行いました。解析領域内のスプラウトのみを定量化しました(1–4)。核の定量化には同様の設定を使用しました(5–6)。このセグメンテーションはすべてのz面で繰り返されました。得られたセグメンテーションマスクは結合され、体積測定を作成しました(7–8)。B) スプラウトの3D成長を定量化した棒グラフ。アッセイは三重測定で実施し、エラーバーは標準偏差(STDEV)を示します。
結論
- 私たちは血管新生のOoCモデルに対する自動化ワークフローを開発しました。
- 自動化の利点には、アッセイのスループットを向上させる能力と、データ再現性の改善が含まれます。
- 血管新生スプラウトの3Dイメージングと解析は、複雑な生物学的プロセスを定量的に記述する手段を提供します。
PDF版(英語)