Application Note MetaXpress 6.5によるカーブフィッティング解析で
実験結果を定量評価
- カーブフィッティングの選択肢が豊富で、最適なグラフ作成が可能
- プレート取得設定が一元化され、操作が簡便
- プレートレイアウトが常に表示・アクセス可能で、視認性が向上
はじめに
Matthew Hammer|アプリケーションサイエンティスト|モレキュラーデバイス
MetaXpress® 6.5(MX 6.5)ハイコンテント画像取得・解析ソフトウェアに搭載された新しいカーブフィッティングアルゴリズムは、データの視覚化と二次解析を容易にするために設計されています。このソフトウェアでは、一連のデータポイントに対して最適なカーブを生成することが可能です。4パラメータロジスティックなどの複数の関数や、EC₅₀やベンチマークレスポンスなどの多様なカーブ出力に対応しており、データを最も適切に表現するグラフやカーブを作成できます。ここでは、毒性アッセイを例に、化合物のEC₅₀値を定義する用量反応関係の生成方法をご紹介します。
マルチ毒性アッセイ(HeLa細胞を使用)
HeLa細胞を、黒色・透明底の384ウェルマイクロプレートに1ウェルあたり2,000細胞で播種し、37°C、5% CO₂環境下で24時間インキュベートしました。その後、抗がん剤を1:3の段階希釈で4反復処理し、48時間インキュベートしました。細胞はHoechst 33342(8 µM)、MitoTracker Orange(100 nM)、Calcein AM(1.25 µM)を含む染色液で30分間染色し、ImageXpress® Micro Confocal ハイコンテントイメージングシステムでイメージングしました。取得したライブセル画像は、MetaXpress 6.5のMulti-Wavelength Cell Scoringモジュールで解析され、細胞やオブジェクトの総数・平均数、サイズ、面積、強度値、各波長に陽性の細胞割合など、複数の読み出し結果が得られました。これらの結果から、カーブフィッティングを含むデータとグラフが生成されました。
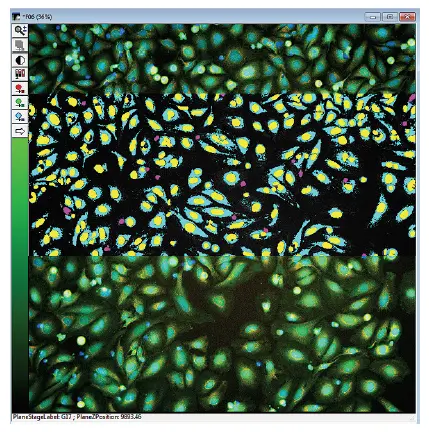
図1. Calcein AMによるライブセル染色の陽性/陰性を示す解析マスク付きイメージング画像 Multi-Wavelength Cell Scoring解析モジュールは、Calcein AMおよびMitoTracker Orange陽性細胞のセグメンテーション、定量、複数の読み出しを行うように設定されています。画像オーバーレイでは、核が青、ライブセルが緑、ミトコンドリアがオレンジで表示されています。ここに示す解析マスクは、波長2(FITC)、Calcein AMの解析結果です。Calcein AM陽性細胞の細胞質マスクは水色、陽性核は黄色、陰性核は紫色で示されています。
データ解析を強化するカーブフィッティング
MX 6.5内でプレートにアノテーションを追加し、細胞ベースアッセイの結果を解釈するための濃度反応曲線を作成しました。Annotate Wellsウィンドウでは、グループタイプ(陰性コントロール、陽性コントロール、テスト)を選択し、ウェルに処理した化合物名、初期濃度、希釈倍率を入力しました。陽性および陰性コントロールはZ値の計算に使用されました。プレートのアノテーションは、データベースまたはローカルファイルとして保存でき、取得済みの特定のプレートに適用可能です
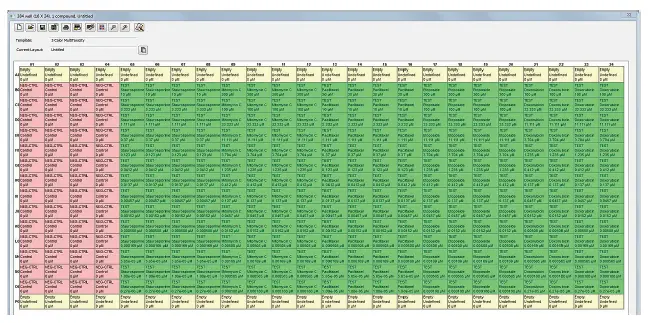
図2. 3色マルチ毒性アッセイ用に設定されたプレートアノテーションファイル MX 6.5のPlate Annotationウィンドウで作成されたプレートには、アッセイに使用された化合物と希釈情報が含まれています。このアノテーションは、カーブフィットを用いた濃度反応曲線の作成に使用されます。
解析済みかつアノテーション済みのプレートからグラフを作成するには、Graphタブに移動し、プロットする測定項目を選択します。この例では、「陽性細胞数 vs. 化合物濃度(μM)」を表示するように設定しました。グラフのデザインが完了したら、「Curve Fit」ボタンを選択して、データに最も適したカーブを追加します。選択可能なフィット関数は十数種類あり、データに最適なものを選べます。今回は用量反応解析を目的として、4パラメータロジスティックカーブを選択し、データポイントの平均値にフィットさせ、「Asymptotic」オプションを有効にし、読み出しとしてEC₅₀を指定しました。
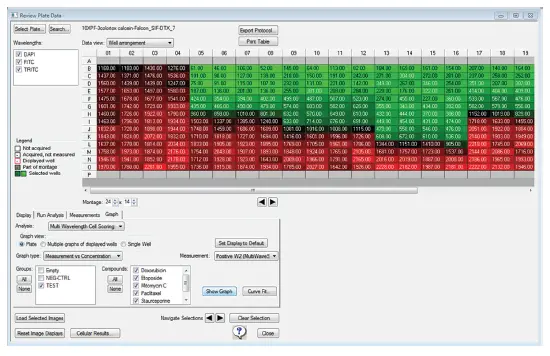
図3. Review Plate Data内でのグラフパラメータ設定 MX 6.5のGraphタブで、グラフタイプ、プロットする測定項目、含めるサンプルを選択します。「Measurement vs. Concentration」を選択すると、プレートアノテーションで設定されたグループと化合物が表示されます。今回プロットに選択した測定項目は、波長2(FITC、Calcein AM)に陽性の細胞総数です。
カーブフィットおよび選択した測定データは、タブ区切りファイルとしてエクスポートでき、グラフは画像ファイルとして簡単に保存可能です。
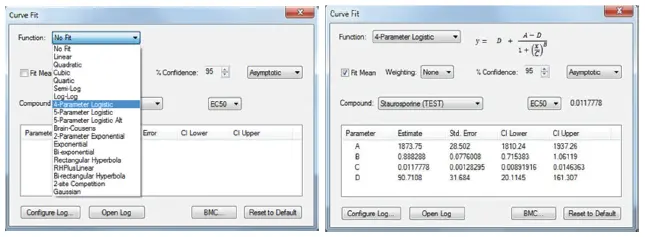
図4. 結果に対応する適切なカーブフィット関数の選択 Curve Fitウィンドウ内では、カーブフィットの設定と、カーブから生成されるデータの最適化が可能です。今回の3色毒性アッセイでは、4パラメータロジスティック関数とEC₅₀読み出しを選択し、用量反応曲線を正確に表現しました。
結論
MetaXpress 6.5ソフトウェアのカーブフィッティングアルゴリズムは、毒性化合物の段階希釈に対するライブセル応答をプロットすることで、その有用性が示されました。プレートへのアノテーション、データのグラフ化、IC₅₀値やベンチマーク濃度などの多様なカーブと読み出しの生成が可能であり、多くの生物学的アッセイにおいて不可欠な機能です。統合されたカーブフィッティング機能により、MX 6.5はイメージングとデータ解析を一体化したオールインワンソフトウェアとして活用できます。
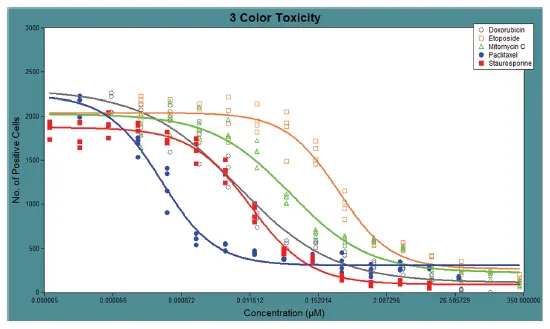
図5. 細胞毒性化合物で処理したHeLa細胞の用量反応曲線 MX 6.5のCurve Fitアルゴリズムによる4パラメータロジスティックフィットにより、化合物の用量反応曲線が適切に表現されました。各化合物のEC₅₀値は以下の通りです。Staurosporine:0.0118 µM、Mitomycin C:0.0527 µM、Paclitaxel:0.0004 µM、Etoposide:0.321 µM、Doxorubicin:0.0073 µM