Application Note AIを活用したラベルフリー腸オルガノイドの表現型解析
- ステップごとのガイド付きワークフローによる簡易化されたハイコンテントイメージング
- SINAPでディープラーニングモデルを作成し、ラベルフリー腸オルガノイドをセグメント化します
- ユーザーフレンドリーなワークフローに従って、セグメント化されたオルガノイドをグループ化する分類ルールを作成します
PDF版(英語)
Zhisong Tong | Research Scientist | モレキュラーデバイス
Prathyushakrishna Macha | Research Scientist | モレキュラーデバイス
はじめに
スフェロイドやオルガノイドといった3Dモデルは、単層の2D培養モデルと比較して生体内環境をよりよく再現できるため、多くの研究分野で注目を集めています。近年のオルガノイド培養技術の進歩により、誘導多能性幹細胞(iPSC)や成体幹細胞を用いて、3Dで臓器様構造を成長させることが可能になりました。小腸は高いターンオーバーを持つ急速に更新される組織です。その主要構成要素は、陰窩(クリプト)と絨毛(ビラス)という主要単位が交互に並ぶ単一の上皮層に存在します(図1)。これらの陰窩には幹細胞が含まれており、分裂して前駆細胞を生じます。したがって、陰窩は腸オルガノイドにおいて重要な部分であり、成熟したオルガノイドはより複雑で多数の陰窩構造を持ちます。
オルガノイドの応用分野には、ハイコンテントスクリーニングを用いた創薬や再生医療などがあります。本ノートでは、ImageXpress® Pico 自動細胞イメージングシステムと CellReporterXpress 自動画像取得・解析ソフトウェアを使用して腸オルガノイドをイメージングしました。CellReporterXpressは、さまざまなアプリケーション向けにガイド付きワークフローを備えたプロトコールを提供することで、ハイコンテントイメージングの複雑さを軽減します。たとえば、新規プレート取得やステッチプレート取得は、複数画像のステッチ有無に応じてよく使用されるプロトコールです。ステップごとのガイド付きワークフローにより、イメージング初心者でも容易に取得プロトコールを設定できます。カスタマイズした設定を保存すれば、後でワンクリックでイメージングを実行できます。
CellReporterXpressの使いやすさは画像取得にとどまりません。強力な画像解析ソフトウェアはオンザフライ解析が可能です。ユーザーは、セルカウント、生死解析、神経突起トレーシング、血管新生アッセイなど、一般的な画像解析ツールを含む25種類の事前構成済み解析プロトコールから選択できます。さらに、IN Carta® 画像解析ソフトウェアのAI対応ツールを使用して高度な画像解析を行うことも可能です。IN Cartaには、核、細胞、点状構造、繊維、神経突起などの一般的な生物構造のセグメンテーション用アルゴリズムが組み込まれており、オプションモジュールのSINAPを利用すれば、ディープラーニングベースのセグメンテーションツールでカスタムニューラルネットワークモデルを作成できます。さらに、IN Cartaには2種類の分類ツールがあります。ユーザー定義の測定値に基づく手動決定木分類器と、機械学習アルゴリズムに基づくPhenoglyphs分類器です。
本テクニカルノートでは、オルガノイドの表現型や形態の変化を記述・測定するためのイメージングおよび画像解析手法の一般的なアプローチを紹介します。これらの方法は、由来や培養プロトコールに関係なく、あらゆるオルガノイドに適用可能です。さらに、ImageXpress Pico 自動細胞イメージングシステムと CellReporterXpress、IN Carta画像解析ソフトウェアを組み合わせた腸オルガノイドのイメージングと解析のワークフローを提示します。特にIN Cartaでは、SINAPによる画像セグメンテーションの後にPhenoglyphsによる分類を行いました。このアプローチを用いて、腸オルガノイドを未成熟、中間、成熟のクラスに分類する簡単な方法を示します。このAI対応分類アプローチは、培養中の腸オルガノイドの成長と発達をモニタリングするために利用できます。
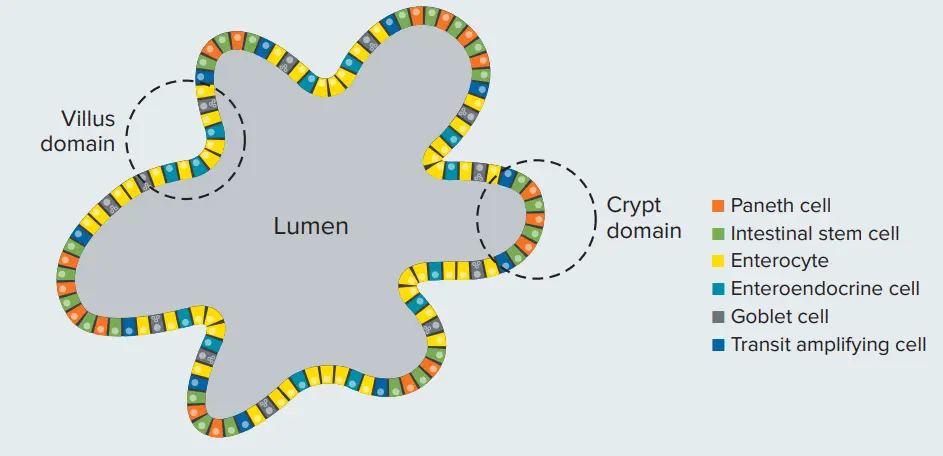
図1. 腸オルガノイドの構造を示す2Dイラスト
方法
オルガノイドイメージング
オルガノイドイメージングの一般的なワークフローを図2に示します。オルガノイドの画像は、ImageXpress PicoシステムでCellReporterXpressを使用し、透過光(TL)で取得しました。Zスタック画像の2Dプロジェクションは、10X対物レンズ、10µmフォーカスステップ、ウェル底面オートフォーカスで取得しました。フォーカスオフセットはCellReporterXpressによって自動的に決定されました。
画像解析
画像はCellReporterXpressからIN Cartaソフトウェア互換フォーマットにエクスポートされました。SINAP(ディープラーニングベースのセグメンテーションモジュール)を使用して、複数画像を含むトレーニングセットで堅牢なカスタムモデルを作成しました。この新しくトレーニングされたモデルを用いて、以降すべての画像を解析しました。Phenoglyphs(機械学習ベースの分類ツール)を使用して、取得したすべてのオルガノイドを未成熟、中間、成熟のクラスに分類しました。
結果
CellReporterXpressによる3Dスタック画像の2Dプロジェクション
マウス腸オルガノイドの画像は、ステッチプレート取得プロトコールで撮影され、ウェル内のすべてのサイトからステッチ画像が生成されました。腸オルガノイドの3D構造のため、Zスタックモードを選択し、オルガノイド全体をカバーする適切なZプレーンを選びました。CellReporterXpressは透過光チャンネル用に異なるZプロジェクションオプション(平均、ベストプレーン、小さなオブジェクトのベストフォーカス、大きなオブジェクトのベストフォーカス)を提供します。平均Zプロジェクションはすべてのプレーンの強度を平均し、ベストプレーンは最もフォーカスの良い1枚を選びます。他の2つは画像を小さなゾーンに分割し、各ゾーンで最適なフォーカスを見つけます。本研究ではTLチャンネルで4つの方式を比較しました。他のイメージャーではベストフォーカスが推奨されますが、陰窩の深さがランダムなため、大きなオブジェクトと小さなオブジェクトのベストフォーカス方式は平均やベストプレーン方式よりも優れた画像品質を提供しました(図3)。そのため、計算負荷が少ない大きなオブジェクトのベストフォーカス方式を採用しました。類似の複雑な構造には、このTL用2Dプロジェクション方式を推奨します。
取得したTLチャンネルの2Dプロジェクション画像は、その後IN Cartaにエクスポートされ、取得中または取得後に解析できます。エクスポートされた画像サイズは、ステッチ画像が2000×2000ピクセルのグリッドに分割され、IN Cartaの要件に合わせて自動調整されました。
腸オルガノイドと陰窩のディープラーニングモデルの生成
ラベルフリーイメージングは、蛍光ラベルを使用せずに生物学的プロセスを研究する重要な手法です。しかし、明視野顕微鏡で取得した画像の自動解析は、高い背景、シェーディング効果、デブリによるアーティファクトなどの要因で困難です。これらの問題を克服するため、IN CartaのディープラーニングベースのセグメンテーションツールであるSINAPを使用しました。
SINAPを用いて、腸オルガノイド解析のための2つのモデルを開発しました。まず、オルガノイド全体を識別するモデルをトレーニングしました。トレーニングセットは、SINAPインターフェース内のブラシとポリゴンツールを使用してオルガノイドと背景領域を手動でラベル付けして作成しました。オルガノイドと背景のコントラストが良好だったため、気道オルガノイド用に作成された既存モデルを微調整することで、14枚のアノテーション画像でモデルをトレーニングできました(図4A)。次に、オルガノイドの陰窩や突起のみをセグメント化するモデルをトレーニングしました。この場合はより慎重なアノテーションが必要で、23枚のトレーニング例で最初のモデルを微調整してモデルを作成しました(図4B)。形状、強度分布、局所的なコンテキストが、オルガノイドのコアを含めずに陰窩を堅牢にセグメント化するための重要な要素でした。モデルのトレーニング後、オルガノイド全体をプライマリターゲット、陰窩をオルガネラターゲットとして設定し、各オルガノイド内の陰窩のリンクとレポートを可能にしました(図4C、図5A)。
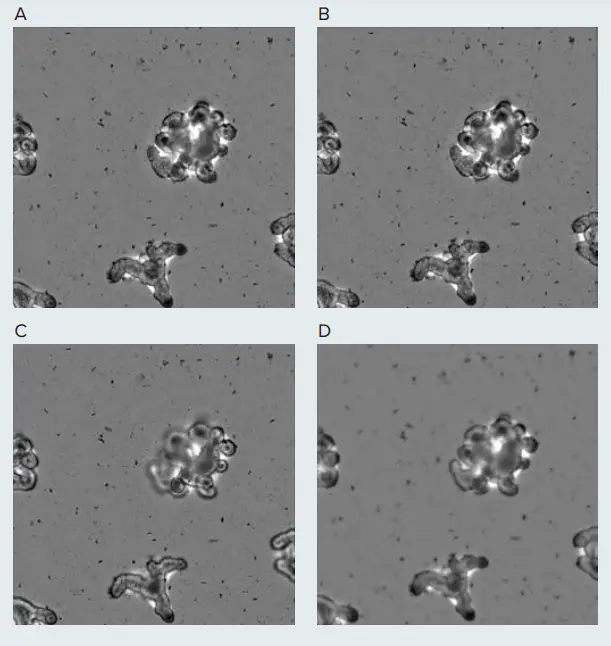
図3. 腸オルガノイドの透過光画像と2Dプロジェクション (A) 大きなオブジェクトのベストフォーカス (B) 小さなオブジェクトのベストフォーカス (C) ベストプレーン (D) 平均
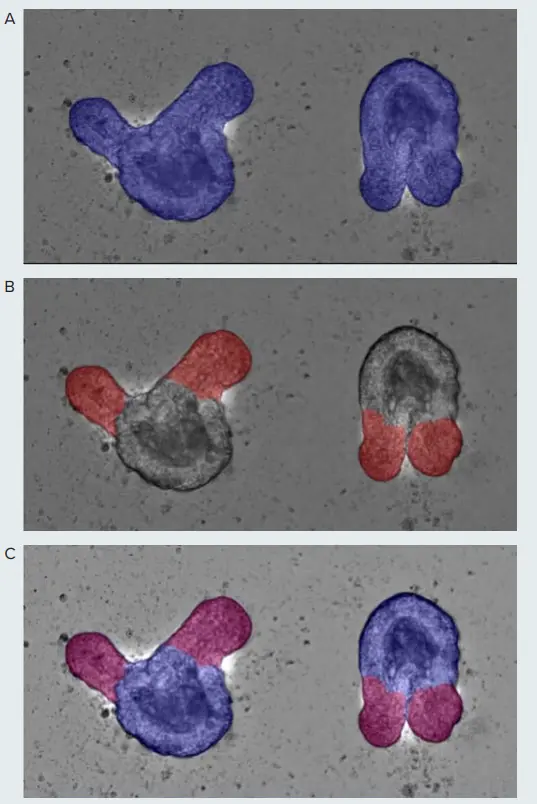
図4. SINAPモデルは、オルガノイド全体(A)と陰窩(B)のマスクをアノテーションするために生成されます。 (C) は2つのマスクの重なりを示します。モデルでは、オルガノイド全体をプライマリターゲット、陰窩をオルガネラとして設定しました。
PhenoglyphsとClassifierによる未成熟、中間、成熟オルガノイドの分類
腸オルガノイドの成長と成熟は、明視野顕微鏡でモニタリングできます。通常、培養3~5日後にオルガノイドは伸長を始め、7日目にはより成熟した複雑な多葉構造(複数のバッドを持つ)が観察されます。培養中の成熟および未成熟腸オルガノイドの数を定量化するため、Phenoglyphsという機械学習ツールを用いて、表現型に基づきオルガノイドを分類しました。
Phenoglyphsでは、形態、相対位置、強度、テクスチャなど、幅広い測定値から選択できます。分類ルールを定義するため、まず専用の機械学習アルゴリズムで表現型クラスターの予備セットを特定しました。次に、未成熟、中間、成熟という目的の表現型を表すクラスターを選択しました。各クラスター内の不均一性を減らすため、過剰な数のクラスターを設定することが推奨されます。本研究では、3つの表現型分類を想定していましたが、最初に20クラスターを要求しました。
測定前に、陰窩関連の測定値が分類に最も寄与すると予想していました。複雑さは陰窩の数を意味するためです。予想通り、これらの測定値は未成熟、中間、成熟オルガノイドを分類する重要な指標としてランク付けされました(図5B)。その後、上位20の測定値を用いてモデルをトレーニングし、その結果を表1に示しました。相関閾値や上位Nは必要に応じて変更できます。推奨としては、選択する測定値の数を 4 × L(Lはクラス数)に近づけ、相関閾値を90に設定して冗長な測定値を排除し、現在のデータセットに過度に特化したモデルの生成を避けます。オルガノイドの代表画像は図5Dに示します。
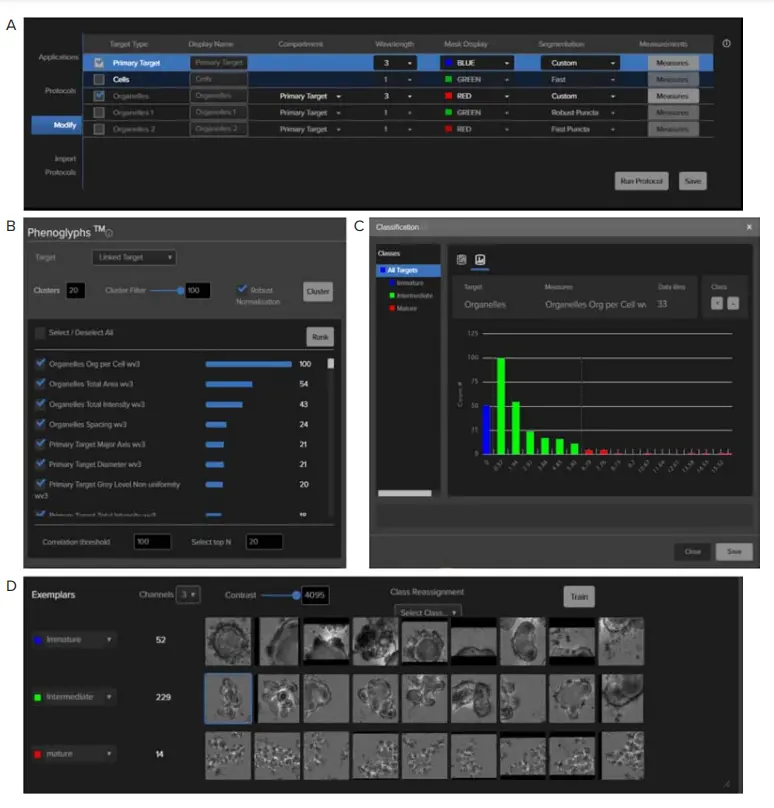
図5. (A) オルガノイド全体モデルをプライマリターゲット、陰窩モデルをオルガネラの1つとして設定 (B) ランキングスコアは、オブジェクトを指定クラスに分ける際の各測定値の重要度を示します (C) Classifierツールで定義された単一測定値(オルガノイドあたりの陰窩数)に基づくルール (D) Phenoglyphsでユーザーに提示されるサンプル画像。クラス定義や誤分類オブジェクトの修正に使用
また、Classifierは、個々の測定値に依存し、上下限をユーザーが手動で定義する、よりシンプルで直接的な分類に使用できます。この場合、オルガノイドあたりの陰窩数(organelles org per cell)を測定値としてクラスを指定しました。図5Cでは、陰窩が0のオルガノイドを未成熟、陰窩が1~6のオルガノイドを中間と分類しました。これはPhenoglyphsで生成された統計(表1)に近い結果です。2つ以上の特徴が分類を支配する複雑なシナリオでは、Phenoglyphsが推奨されます。ウェル別、視野別、クラス別の集計結果や単一ターゲットデータは.csvファイルとしてエクスポートでき、さらなる解析に利用できます。
| フェノグプリフス | 分類 | |
|---|---|---|
| 未熟 | 52L | 51 |
| 中間 | 229L | 231 |
| 成熟 | 14 | 13 |
表1. PhenoglyphsとClassifierに基づく分類結果
結論
- ImageXpress Picoシステムは、ガイド付きワークフローによる迅速で便利なハイコンテントイメージングを可能にします。
- SINAPは、従来の画像解析アルゴリズムでは解析が難しいオブジェクトをセグメント化できる強力なツールです。
- Phenoglyphsは、ラベルフリーオルガノイドの堅牢な分類を可能にします。
PDF版(英語)
