Application Note 細菌増殖アッセイのキネティック解析と応用
- Workflow Editorを使用して、細胞密度と蛍光タンパク質のキネティックトレースを同時に検出し、時間を節約できます
- 事前構成済みかつカスタマイズ可能なデータ解析オプションにより、データレポートの作成を簡素化できます
- 必要なデータ出力パラメータを表形式またはプレート形式に容易に統合し、散布図や棒グラフとしてグラフ化できます
PDF版(英語)
はじめに
Michel Hoogenkamp|研究技術者|アムステルダム歯科学術センター(ACTA)
Cathleen Salomo|フィールドアプリケーションサイエンティスト|モレキュラーデバイス
殺菌が困難な細菌は、院内感染において問題となっています。これらの細菌を殺す化合物を特定することは、多くの製薬企業にとって関心のある課題です。これらの化合物の有効性をスクリーニングするためのアッセイを開発し、最適化することは、多くの微生物学者が直面する課題です。
ここでは、SoftMax® Pro 7.0(またはそれ以降)のデータ取得・解析ソフトウェアを使用した細菌増殖アッセイのセットアップについて説明します。Workflow Editorの取得機能を使用して、細胞密度とGFPシグナルの両方をタイムコースで記録しました。さらに、GFPシグナルを細胞密度に正規化する方法や、成長速度などのキネティック関連情報を抽出する方法など、ソフトウェア内のさまざまなデータ変換ステップについても解説します。
材料
- エンテロコッカス・フェカリス株OG1RF 下記が含まれます:
◦プラスミドpMV158-GFP(緑色蛍光タンパク質)
- 96ウェル ブラック・ウォールドμClearマイクロプレート(Greiner Bio-One)
- qPCRシール(Eurogentec)
- SpectraMax i3x マルチモードマイクロプレートリーダー(モレキュラーデバイス):
◦SoftMax Pro 7.0(またはそれ以上)ソフトウェア
方法
Workflow Editorを使用したデータ取得
細菌増殖データは、オランダのAcademic Centre for Dentistry(ACTA)の予防歯科学部門において、SpectraMax® i3xマルチモードマイクロプレートリーダーを使用して取得しました。ここでは、プラスミドpMV158-GFPを含むEnterococcus faecalis株OG1RFをさまざまな増殖条件で使用しました。GFP(緑色蛍光タンパク質)の発現は異種発現であり、この株はHoogenkampらの論文¹で記載されており、GFPは染色が困難な種E. faecalisの生存性マーカーとして研究されています。150 μLのE. faecalisをPBSに懸濁し、PBSをバックグラウンドコントロールとして、96ウェルの黒色壁μclearプレートに分注しました。蒸発を防ぐため、マイクロプレートはqPCRシールで覆いました。その後、マイクロプレートをSpectraMax i3xプレートリーダーにセットし、キネティック測定の間、37°Cでインキュベートしました。
SoftMax Pro Workflow Editorを使用して、吸光度測定(PlateOD₆₀₀)と蛍光測定(PlateGFPBottom)を含むキネティックサイクルを作成し、測定間に5秒間の線形プレートシェイクを設定しました(図 1)。吸光度は600 nm(OD₆₀₀)で測定し、細菌の増殖を判定しました。GFP発現(生存性の指標)は、励起485 nm(帯域幅9 nm)、蛍光515 nm(帯域幅15 nm)のボトムリード蛍光検出でモニターしました。両方のデータトレース(吸光度と蛍光)は、2つの独立したプレートセクションで記録しました。キネティックサイクルは15分ごとに繰り返し、合計18時間測定しました。オプションのソフトウェア機能により、キネティックリードを一時停止・再開でき、測定中に試薬を追加することも可能です(本実験では未使用)。得られたデュアルリードモードのキネティックデータは、SoftMax Proソフトウェアで解析しました。
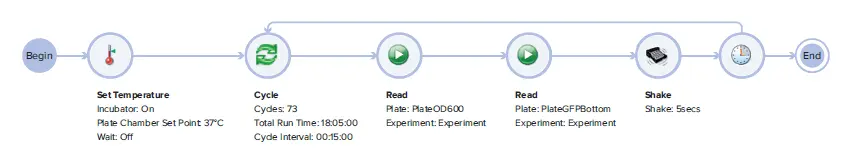
図1. デュアルリードモードのキネティック測定用SoftMax Pro 7 Workflow Editor ドラッグ&ドロップ機能により、簡単に設定でき、カスタマイズされたワークフローを迅速に構築できます。
Reduction Settingsダイアログを使用したデータ解析オプション
データ解析の初期ステップとして、ブランクを定義するかどうかを決定する必要があります。ブランクウェルの位置はテンプレートエディタで設定し、グループブランクまたはプレートブランクを選択できます。プレートブランクは、各タイムポイントでプレート内のすべてのサンプルウェルのローデータから差し引かれ、グループブランクは関連するサンプルウェルのみから差し引かれます。本実験では、バッファーのバックグラウンドトレースをキャプチャし、細菌やその他の種による汚染の可能性をコントロールしました。バッファーはOD₆₀₀やGFP測定に干渉しなかったため、差し引きは不要でした。もし培地やバッファー成分がOD₆₀₀に干渉したり蛍光を発したりする場合は、吸光度または蛍光チャネルの真のシグナル値を取得するためにブランクの差し引きを推奨します。これにより、測定の比較可能性も確保できます。
Reduction Settingsダイアログでは、高度なキネティックデータ解析が可能です。図2は、細菌増殖実験のReduction Settingsを示しています。メニューは「ローデータ」と「データリダクションステップ」の2つのセクションに分かれています。
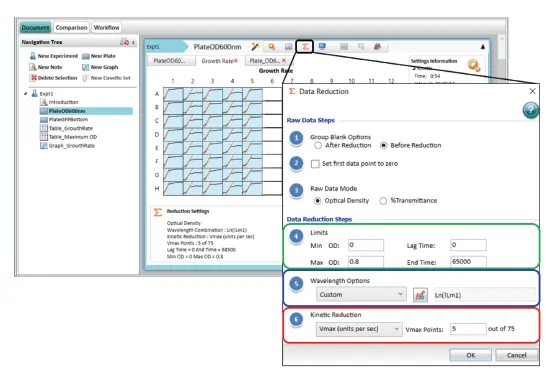
図2. Reduction Settingsメニューのデータ解析オプション この例では、プレートセクション「PlateOD600nm」の光学濃度データの対数変換と、その後のVmaxレート値へのリダクション(最急部分を取得するために5 Vmaxポイントを使用)を示しています。
ローデータステップには、すべてのキネティックトレースの最初のデータポイントをゼロに設定するオプションがあります。さらに、ブランク計算はキネティックリダクションの前または後に含めることができます。「リダクション前」を選択すると、平均化されたブランクウェルのキネティックデータトレースが各タイムポイントで個別にすべてのローデータセットから差し引かれます。「リダクション後」を選択すると、Vmaxレートなどのリダクション後のブランクウェルデータの平均がサンプルウェルデータから差し引かれます。
データリダクションステップとデータ出力タイプには、以下のオプションがあります(図2):
- Limits:必要に応じて、リミットはデータリダクション解析に含めるキネティックデータの範囲を制限します。リミットはシグナル軸(OD、RLU、RFU)や時間軸(秒)に設定できます。同じ解析範囲がすべてのウェルに適用されます(図 2、緑枠)。
- Wavelength Options:選択したリミット内でシグナル軸を変換します。デフォルトでは、最初に測定された波長(!Lm1)が選択されます。ここでは、シグナル軸変換の2つの例を示します:
- 正規化:2つの波長データトレースが同じプレートセクション内で取得された場合、ドロップダウンメニューには、比率(波長1/波長2 = !Lm1/!Lm2)などの事前構成済み選択肢が表示されます。本アプリケーションノートで説明するデータでは、データトレースは2つの独立したプレートセクションで取得されたため、比率シグナル正規化にはカスタム式が必要でした。GFPシグナルを細菌細胞密度に正規化するため、次の式を入力しました:
- 正規化:2つの波長データトレースが同じプレートセクション内で取得された場合、ドロップダウンメニューには、比率(波長1/波長2 = !Lm1/!Lm2)などの事前構成済み選択肢が表示されます。本アプリケーションノートで説明するデータでは、データトレースは2つの独立したプレートセクションで取得されたため、比率シグナル正規化にはカスタム式が必要でした。GFPシグナルを細菌細胞密度に正規化するため、次の式を入力しました:
$$\frac{\text{!KinPlot@PlateGFPBottom}}{\text{!KinPlot@PlateOD₆₀₀}}$$
この式は、いずれかのプレートセクションに適用しました。!KinPlotは、記号「@」とプレートセクション名で参照された指定プレートセクションのキネティックデータセットを抽出します。ローデータトレースと変換後(正規化後)のデータトレースの比較ビューは図3に示されています。
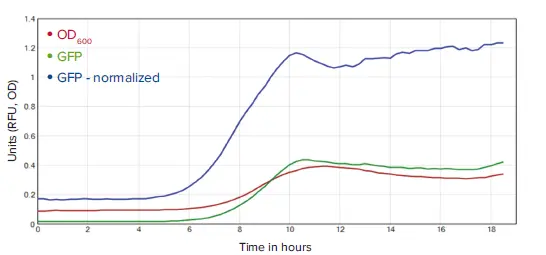
図3. GFPシグナルをOD₆₀₀で細胞密度に正規化した結果
-
- OD₆₀₀データトレースの指数増殖期を線形化するため、OD₆₀₀プレートセクションに対して対数変換を適用しました
- カスタム式として自然対数を使用し、Ln(!Lm1)を入力しました(図2の青枠参照)
- ローデータと対数スケーリング後のデータ比較は図 4に示されています
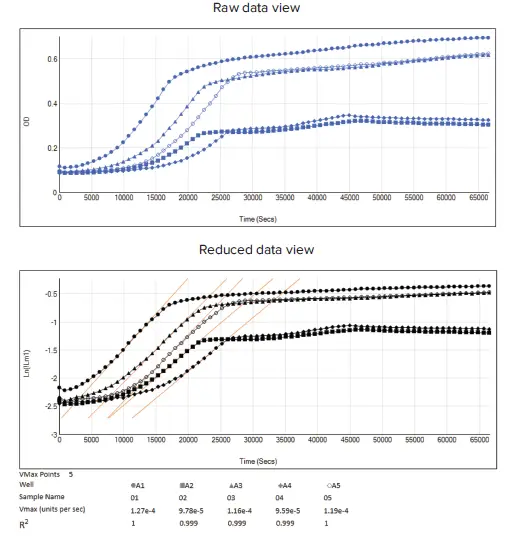
図4. OD₆₀₀データに対数スケーリングを適用したローデータとリダクション後データのビュー ウェルA1からA5は、各異なる実験条件の例として使用しました。上段:OD₆₀₀キネティックデータトレースのローデータ(青線)。下段:対数スケーリングを適用した変換後のローデータ(黒線)と、Vmaxデータリダクションを適用して指数増殖期の成長速度を決定した結果(オレンジ線)。対応するリダクション設定は図2に示されています。
キネティックリダクション:このステップでは、各データトレースをVmaxレートやオンセットタイムなどの単一値に変換します。利用可能な事前構成済みキネティックリダクションパラメータの完全なリストと詳細は表 1に記載されています。上記の対数スケーリングの例を用いて、キネティックリダクションを説明します:
| キネティックリダクション | 詳細 |
|---|---|
| Vmax | キネティックトレースの最大傾きで、単位はミリユニット/分またはユニット/秒です。Vmaxポイントの数は、傾きを決定するために使用される線分の最大サイズを定義します。 |
| Time to Vmax | Vmaxに到達するまでの経過時間データは、凝固化学などのアプリケーションに有用です。試薬濃度の変化はVmaxを変えず、反応が最大速度に達する時間を変えます。 |
| Slope | Vmaxレートと同じ(単位/秒)で、リダクションリミット内のすべてのデータポイントを使用して傾きを決定します。 |
| Onset time | 非線形キネティック反応を解析する方法です。オンセットタイムは、指定されたODまたはRFU/RLU値に到達するまでに必要な時間を報告します(オンセットOD/RFU/RLU)。エンドトキシン試験などのカスケード反応に有用です。 |
| Time at minimum or maximum | リダクションリミット内で最小または最大のOD、RFU/RLU、または%Tに達した時点を報告します。 |
| Time at 1/2 maximum | リダクションリミット内で最大値の半分に達した時点を報告します。 |
| Area under curve | このリダクションは、リダクションリミット内で定義されたデータポイントに基づいて曲線下の面積を推定します。データプロットは、連続するデータポイントとX軸座標に頂点を持つ台形の系列として扱われ、各台形の面積を計算して合計します。 |
| Minimum or maximum | リダクションリミット内で最小または最大のOD、RFU/RLU、または%Tを報告します。 |
| Max-Min | リダクションリミット内で最大値から最小値を差し引いた値を報告します。 |
| Mean | リダクションリミット内で平均OD、RFU/RLU、または%Tを報告します。 |
表1. 事前構成済みキネティックリダクションオプション
OD₆₀₀データトレースの対数変換の次のステップとして、図 2(赤枠)に示すように、Vmaxレートを抽出することで最大成長速度を取得できます。Vmaxレートは、傾きを決定するために使用される線分の最大サイズを定義するVmaxポイントをユーザーが調整できるという利点があります。Vmaxは、キネティックリダクション後のプロットでオレンジ色の線として表示されます(図 4、リダクション後データビュー)。 プレートセクションビューで全ウェルのリダクション後キネティックグラフを最適に比較するには、[Display]に移動し、[Reduced Data]オプションで[Plot]を選択します。
異なる増殖条件を数値結果として表形式で比較するために、テンプレートエディタツールを使用してウェルをサンプルグループに割り当て、図 5(上段)に示すようにリダクション後データ(Vmaxレート)を表で表示します。後続の計算ステップにより、成長速度(k)と倍加時間(g)の両方が取得されます。データの視覚化を改善するために、結果は棒グラフとして表示できます(図 5、下段)。これにより、増殖条件や処理効果をより容易に評価できます。
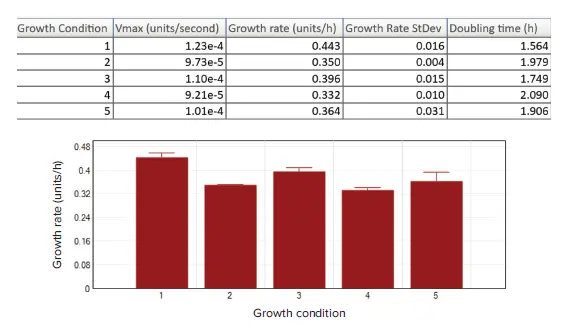
図 5. 結果表示オプション プレートセクションのリダクション後データ表示と、E. faecalisの各実験条件の結果(カラム1~5、n=8)は、結果テーブルおよび棒グラフとして要約されています。Vmaxレートは、成長速度(k = Vmax × 3600)および倍加時間(g = ln2 / k)の両方を取得するために使用されました。
結論
SoftMax Pro 7ソフトウェアのWorkflow Editorは、SpectraMax i3xプレートリーダーを含むモレキュラーデバイスのマルチモードマイクロプレートリーダーと組み合わせることで、光学濃度と蛍光タンパク質発現の両方を同時に記録するデュアルリードモードのキネティック増殖データ取得を柔軟に実現します。解析はSoftMax Proソフトウェアに組み込まれており、データ正規化や対数スケーリング調整など、細菌増殖データに対するさまざまなデータ解析オプションと変換を提供します。Vmaxレートやオンセットタイムなど、さまざまなキネティックデータリダクションパラメータはソフトウェアに事前構成されています。必要なデータ出力パラメータは、表形式またはプレート形式に容易に統合でき、棒グラフや散布図としてグラフ化することで、さまざまな実験条件の評価をサポートします。
参考文献
- Hoogenkamp MA, Crielaard W, Krom BP. Uses and limitations of green fluorescent protein as a viability marker in Enterococcus faecalis: An observational investigation. J. Microbiol. Methods (2015) Aug;115:57-63.
PDF版(英語)