アプリケーションノート
NanoBRET システムによる p53 とMDM2 のタンパク質間相互作用の測定
利点
- 高輝度のNanoLuc によるシグナルと大きなスペクトル分離により、他のBRET 技術よりも感度が改善
- 生細胞内における生理学的レベルのタンパク質相互作用を高感度に検出
- SoftMax Pro ソフトウェアによるNanoBRET 比の自動計算と結果のグラフ化
SpectraMax iD5マルチモードマイクロプレートリーダーについて問い合わせる
はじめに
BRET(生物発光共鳴エネルギー移動)は、生物発光するドナーと蛍光を発するアクセプターとの相互作用を利用したタンパク質-タンパク質間またはタンパク質- リガンド間の相互作用を測定するための技術です。ドナーとアクセプターが互いに10 nm より近接したとき、ドナーはアクセプターを励起し、次いでアクセプターから蛍光が発せられます。対象のタンパク質をドナーとして、その結合パートナーをアクセプターとしてそれぞれタグ付けすると、ドナーおよびアクセプターから発せられた光をマイクロプレートリーダーで検出することにより、タンパク質間相互作用を測定できます。
Promega社のNanoBRET™ テクノロジーは、高輝度な発光ドナー(NanoLuc ルシフェラーゼ)、最適化されたエネルギーアクセプター(HaloTag®-NCT)、そしてドナーとアクセプターの間の大きな波長差による分離を巧みに組み合わせることにより、BRET1やBRET2といった従来のBRET 法よりも改良された技術です(図1)。これらの改善は、高いシグナルと低いバックグラウンド、そして感度の向上をもたらし、生細胞内におけるタンパク質間相互作用の検出を可能にします1。
NanoBRET シグナルの検出と得られたデータの解析には、高感度な測定装置と高度なソフトウェアが必要です。SoftMax® Pro ソフトウェアを搭載したSpectraMax® iD5 マルチモードマイクロプレートリーダーにより、最適化されたフィルターセットを使用してNanoBRET のデータを取得し、カーブフィッティングを含む解析を迅速に行うことができます。このアプリケーションノートでは、相互作用する2 つのタンパク質p53 とMDM2 から成るNanoBRET™ PPI Control Pair を用いた、SpectraMax iD5 マイクロプレートリーダーの評価について説明します。
p53 経路の活性化因子であるNutlin-3 を用いてp53 とMDM2の相互作用を濃度依存的に阻害し、その結果をSoftMax Pro ソフトウェアを用いて解析し、グラフを作成しました。
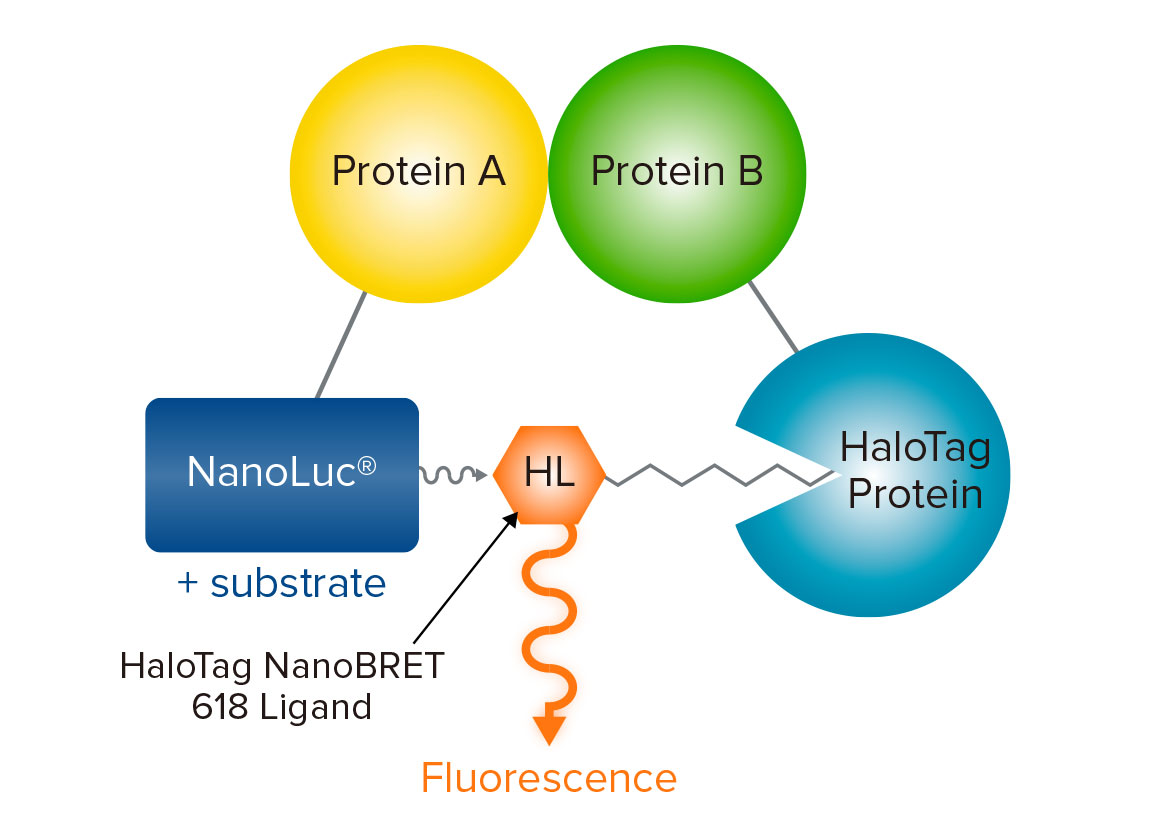
図1 NanoBRET アッセイ。
NanoLuc-Protein A 融合タンパク質(エネルギー供与体、ドナー)が蛍光標識HaloTag-Protein B 融合タンパク質(エネルギー受容体、アクセプター)と相互作用すると、ドナーとアクセプターが接近し、エネルギーが転移します。
材料
- NanoBRET PPI Control Pair(p53、MDM2)、Promega、Cat# N1641
- NanoBRET Nano-Glo® Detection System、Promega、Cat# N1661
- ViaFect™ Transfection Reagent、Promega、Cat# E4981
- Nutlin-3、Millipore-Sigma、Cat#6287
- Opti-MEM™ Reduced Serum Medium, No phenol red、ThermoFisher、Cat#11058021
- HEK-293 細胞、ATCC、Cat# CRL-1573
- Eagle’s Minimum Essential Medium (EMEM)、Corning、Cat#10-010-CV
- BenchMark™ Fetal Bovine Serum、Gemini Bio-Products、Cat#100-106
- ペニシリン- ストレプトマイシン(10000 U/mL)、ThermoFisher、Cat#15140122
- 6 ウェルクリアマイクロプレート、VWR、Cat#1006-892
- 96 ウェル白色平底ポリスチレンマイクロプレート、Corning、Cat#3917
- SpectraMax iD5 マルチモードマイクロプレートリーダー、Molecular Devices、Cat#iD5-STD
方法
HEK-293 細胞を細胞培養培地(EMEM + 10% ウシ胎児血清(FBS)+ 1% ペニシリン/ ストレプトマイシン)を用いて4× 105cells/mL の細胞懸濁液を調整し、6 ウェルプレートにウェル当たり2 mL(8× 105 細胞)となるように加えました。細胞を37℃、5% CO2 環境下で4 ~ 6 時間培養し、ウェル底に付着させました。次に、2 μg のp53-HaloTag 融合ベクターDNA および0.2 μg のNanoLuc-MDM2 融合ベクターDNA をViaFect Transfection Reagent:DNA 比が3:1 となるように、最終容量100 μL のOpti-MEM Reduced Serum Medium を用いて調製し、トランスフェクションを行いました。さらに、細胞を37℃、5% CO2 環境下で20 ~ 24 時間インキュベートしました。
HEK-293 細胞を1,000 rpm で5 分間遠心し回収しました。
Opti-MEM + 4% FBS を用いて細胞密度が2.2 × 105 cells/mLとなるように調整し、2 本の15 mL コニカルチューブに分注しました。一方のチューブを0.1 μM HaloTag 618 Ligand で処理し、他方をリガンドなしとして処理しました。細胞を96 ウェル白色マイクロプレートに2 × 104 cells/well となるように加えて、直ちに、1:3 に段階希釈したNutlin-3(各濃度n = 4)あるいは0.5%DMSOで処理しました。細胞を37℃、5% CO2 環境下で一晩インキュベートしました。
NanoBRET Nano-Glo 基質の5 倍溶液をOpti-MEM + 4%FBSを用いて調製し、それを25 μL ずつ各ウェルに添加しました。
ドナーの発光(447 nm)とアクセプターの蛍光(610 nm)を、SpectraMax iD5 マイクロプレートリーダーを用いて表1 に示す設定で測定しました。SoftMax Pro ソフトウェアにおいて、アクセプターシグナルをドナーシグナルで割ることによってNanoBRET 比を計算し、その比に1000 を乗じて、整数ミリBRET 単位(mBU 値)を算出しました。mBU 値は、リガンドを含まないサンプルのmBU値平均値を差し引くことによってバックグラウンドを補正しました。Nutlin-3 処理した細胞の測定結果は、SoftMax Pro ソフトウェア(Ver. 7.0.3 以降)を用いて解析し、4-parameter カーブフィッティングによりプロットしました。
また、アッセイ系の精度を評価するために、Nutlin-3 の各濃度でZ’ Factor を求めました。

表1 SpectraMax iD5 マイクロプレートリーダー用のNanoBRET の検出設定。
結果
NanoBRET 比(mBU)に対するNutlin-3 濃度の関係をSoftMax Pro ソフトウェアを用いて4-parameter カーブフィッティングによりプロットしました(図2)。Nutlin-3 のIC50 値は1.2 μMと求められ、NanoBRET PPI Control Pair においてPromega 社で示されている値と一致しました2。
図2 に示した曲線を形成するNutlin-3 のすべての濃度において、Z’ Factor は0.7 以上と計算され、また、0.07 μM 以下の濃度では、0.9 と計算されました。これらの値は、このNanoBRET アッセイがロバストであることを意味しています。
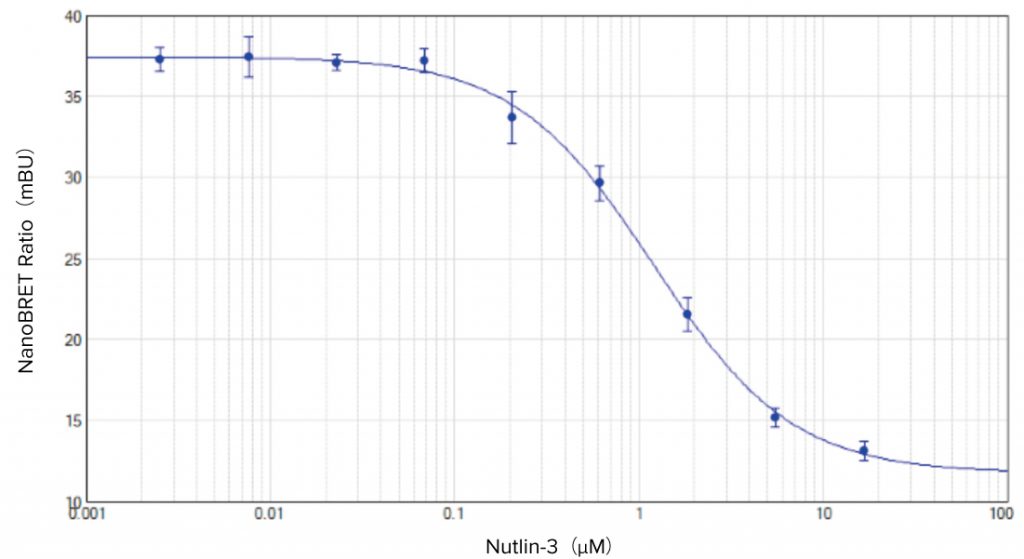
図2 Nutlin-3 によるp53とMDM2 の相互作用の阻害。
SoftMax Pro ソフトウェアの4 パラメーターカーブフィットによって求められたIC50 は1.2 μM(n = 4)であった。
結論
NanoBRET のドナーおよびアクセプターシグナルの検出に最適なフィルターを搭載したSpectraMax iD5 マイクロプレートリーダーとNanoBRET PPI Control Pair(p53、MDM2)を用いて、このアッセイ系におけるSpectraMax iD5 の性能を評価しました。Nutlin-3 を用いて、p53 とMDM2 の相互作用を濃度依存的に阻害した結果、期待されるIC50 値1.2 μM が得られました。
アッセイ系の精度およびレプリケート内のばらつきを評価するために計算したZ’ Factor は、試験したすべてのサンプル濃度において0.7 ~ 1 であり、SpectraMax iD5 マイクロプレートリーダーの感度およびNanoBRET アッセイのロバスト性が確認できました。SoftMax Pro ソフトウェアを使用することで、NanoBRET比の計算とデータのプロットを解析の流れに沿って自動的に行うことができました。
SpectraMax iD5マルチモードマイクロプレートリーダーについて問い合わせる
参考資料
- Machleidt T, Woodroofe CC, Schwinn MK, Mendez J, Robers MB, Zimmerman K, Otto P, Daniels DL, Kirkland TA, and Wood KV. NanoBRET̶A Novel BRET Platform for the Analysis of Protein-Protein Interactions. ACS Chem. Biol. 2015, 10, 1797-1804.
- Technical Manual: NanoBRET Protein: Protein Interaction System. Promega Corporation.
