eBook
核酸とタンパク質をプロレベルに検出
目次
- SpectraMax マイクロプレートリーダーを用いたQuant-iT PicoGreen dsDNAアッセイ
- SpectraMax マイクロプレートリーダーを用いたDNAおよびRNAの吸光度測定
- CyQUANT 細胞増殖アッセイを用いた細胞増殖の測定
- 1cm光路長に自動補正した光学密度測定
- NanoOrangeアッセイを用いたタンパク質定量
- BradfordおよびELISAアッセイを用いたタンパク質定量
- 少量のDNA 試料の蛍光ベースの定量
SpectraMax マイクロプレートリーダーについて問い合わせる
核酸とタンパク質の正確で高感度な検出は、多くの実験に不可欠です。Molecular Devices SpectraMax® マイクロプレートリーダーは、核酸およびタンパク質検出用の多数のアッセイをサポートします。SoftMax® Pro ソフトウェアの解析能力と併せて、読みやすく、ユーザーがカスタマイズしやすいレポート形式で、定量結果を提供します。
SpectraMax マイクロプレートリーダーを用いたQuant-iT PicoGreen dsDNAアッセイ
二本鎖DNA は通常、DNA 溶液の260 nm の吸光度測定により、マイクロプレートリーダーで定量します。しかし、この方法では、通常の吸光度マイクロプレートリーダーで最低250 ng/mL までしか測定できません。サブクローニング用のDNA 断片の精製やDNA 増幅産物の定量などの少量の試料を扱う生物学アプリケーションでは、より感度の高い方法が必要です。
Molecular Probes のQuant-iT PicoGreen dsDNA アッセイは、DNA 特異的で、通常の吸光度法よりも約1000 倍高感度です。マイクロプレート形式でのこのアッセイのダイナミックレンジは、単一色素濃度で250 pg/mL ~ 1000 ng/mL です。
このアプリケーションノートでは、SpectraMax® 蛍光マイクロプレートリーダーとQuant-iT PicoGreen アッセイを用いて、僅か100 pg/mL の二本鎖DNA を高い信頼性で測定できることをご紹介します。
アプリケーションノートをダウンロードする▶
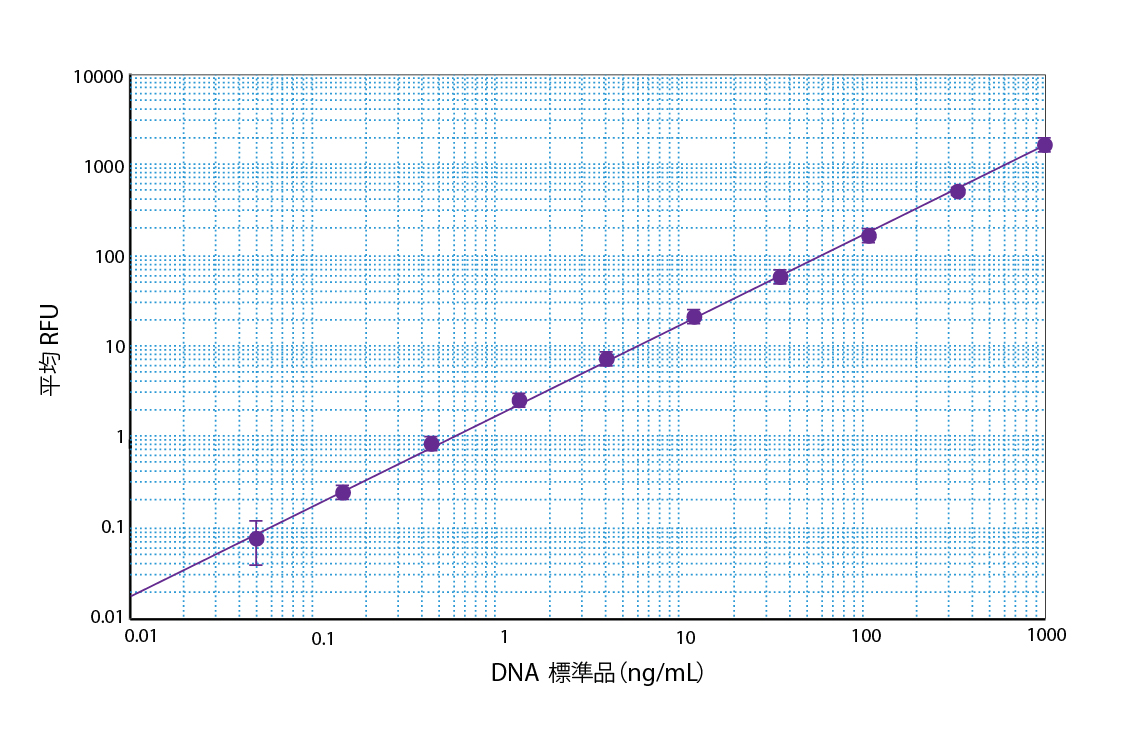
DNA 標準曲線。Quant-iT PicoGreen dsDNAアッセイとSpectraMax M5マイクロプレートリーダーを用いて、50pg/mL ~ 1000 ng/mLのDNA スタンダードをアッセイした。SoftMax® Proソフトウェアでlog-logカーブフィッティングで標準曲線をプロットした(r2 = 1.000)
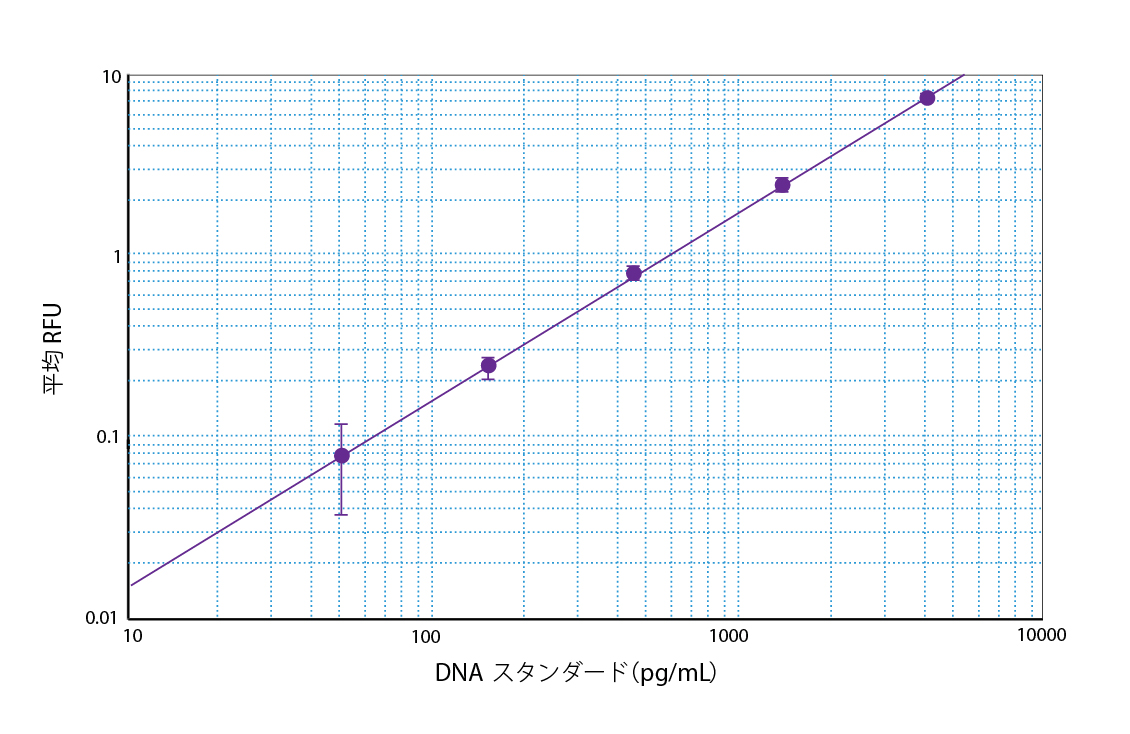
DNA 標準曲線の下端。DNA 標準曲線の下端(50pg/mL ~ 4.1ng/mL)のプロット(r2 = 1.000)。
- 最低63pg/mLまでのDNAの高感度蛍光定量
- 4桁におよぶ線形ダイナミックレンジ
- SoftMax Proソフトウェアのプリセットプロトコルで容易に結果を解析
SpectraMax マイクロプレートリーダーを用いたDNAおよびRNAの吸光度測定
モレキュラーデバイスが初の紫外領域(UV)を測定可能なマイクロプレートリーダーを導入したことで、マイクロプレートの紫外(UV)測定が可能になりました。それ以降、DNA、RNA、タンパク質のマイクロプレート測定は、一般的に広く使用されるようになりました。
しかし、マイクロプレートアッセイでは、マイクロプレート素材の光学特性を知っておく必要があり、特にUV 範囲で正確な吸光度を得るためには、通常のキュベットよりも技術的に多くの注意を払う必要があります。マイクロプレートアッセイに適用困難となる最も多い原因は、このような細かい部分への注意が欠ける場合です。マイクロプレートを用いた吸光度測定は、光路長の変動に影響を受けやすく、空気/液体界面の表面効果の干渉に対して脆弱です。現在のマイクロプレートリーダーは、以前のワイドビームプレートリーダーよりも細い光ビームを使用しているため、ほこりの影響を受けて測定に誤りが生じることがあります。測定時の光ビーム内の粒子は、最高0.3 OD の不自然な吸光度スパイクを引き起こすことがあります。したがって、試料溶液に粒子が存在しないことが特に重要です。マイクロプレートで正確かつ再現可能な吸光度の測定結果を得るためには、前述の全ての因子に留意しなければなりません。
ここでは、SpectraMax® マイクロプレートリーダーでDNA/RNA 吸光度測定を最適化するためのガイドラインをご紹介します。
アプリケーションノートをダウンロードする▶
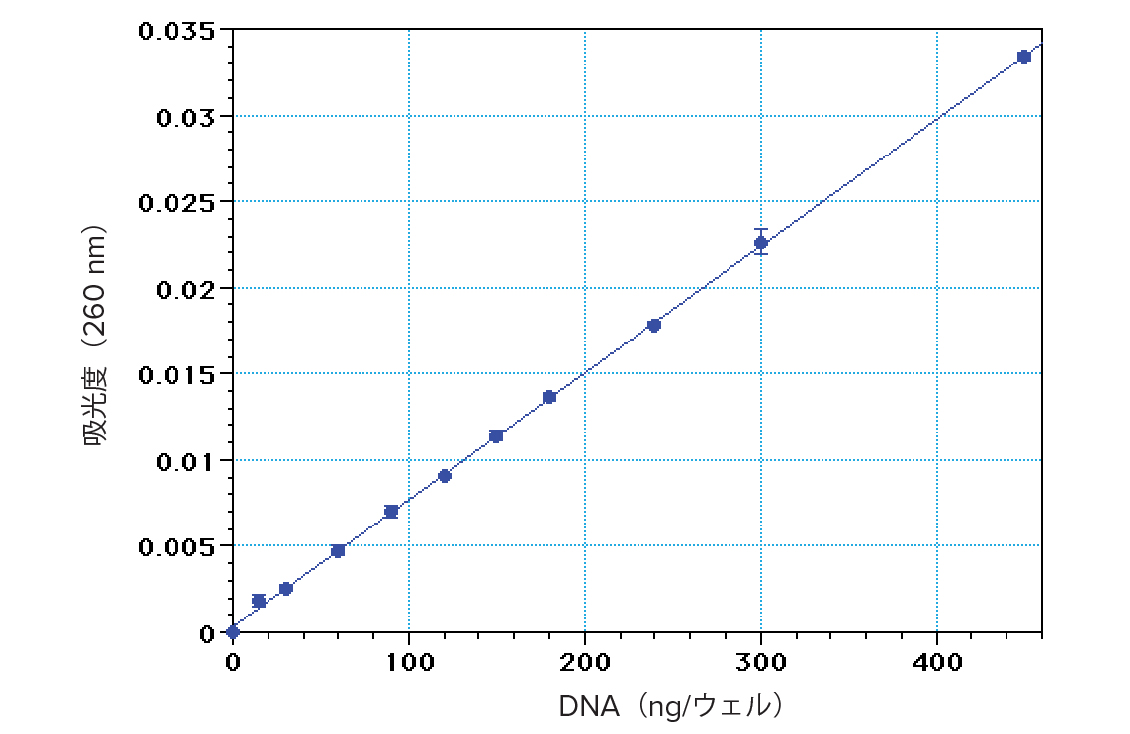
DNA 標準曲線。水に溶解した仔ウシ胸腺DNA で作成した標準曲線。
Costar UV マイクロプレートに300 μL を3 ウェルにそれぞれ分注し、PathCheck を適用して260 nm で測定した。
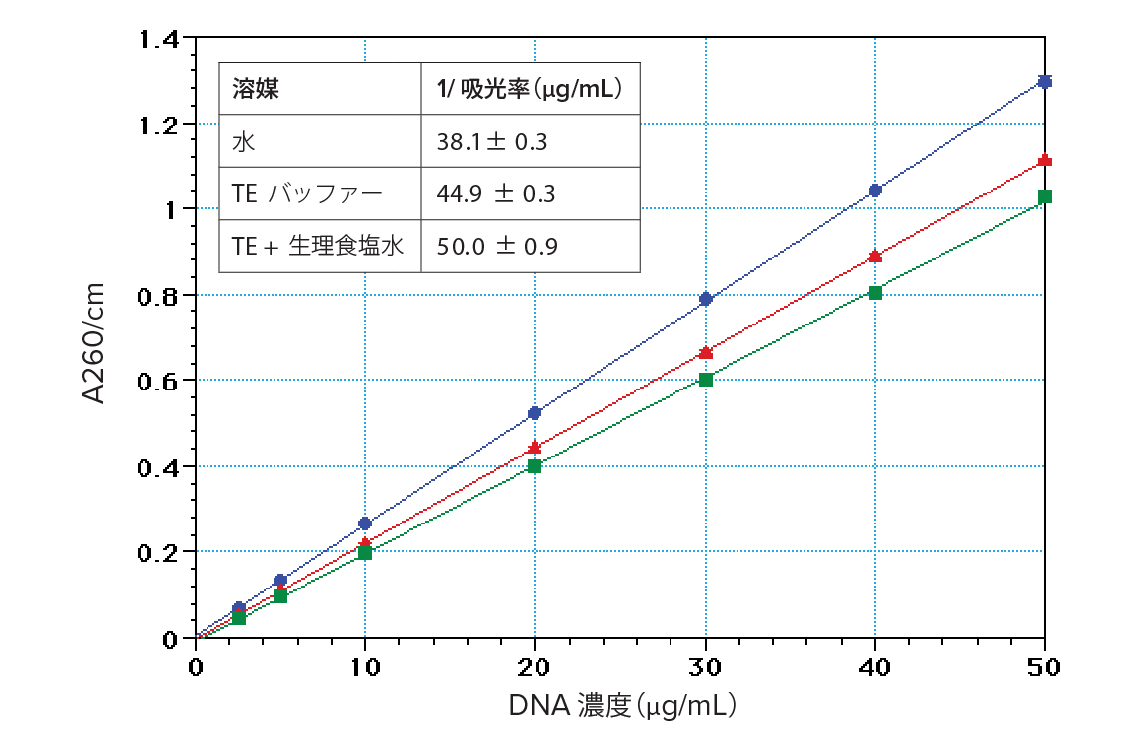
吸光率を用いたDNA/RNA 推定。260 nm のDNA 吸収に対するイオン強度の影響。DNA(Sigma Type I “Highly polymerized”, Cat. No. 1501)を脱イオン水またはTE バッファー(10 mM Tris, 1 mM EDTA, pH 7.4)あるいはTES(TE バッファー + 0.9% NaCl)に溶解した。挿図:1/ 吸光率の値:7 つの濃度(2.5 ~ 50 μg/mL)の平均、それぞれ4 回測定。
- 標準曲線不要で核酸を直接定量
- 最低250 ng/mL までDNA 定量可能
- SoftMax Pro ソフトウェアのプリセットプロトコル
CyQUANT細胞増殖アッセイを用いた細胞増殖の測定
蛍光を用いて細胞増殖を定量すると、細胞増殖に対する薬剤やその他の実験的処理の影響を簡単にモニタリングできます。
Life Technologies のCyQUANT細胞増殖アッセイキットは、蛍光マイクロプレートリーダーを用いて細胞増殖を定量する、感度の高い迅速で簡便な方法です。CyQUANT GR色素は細胞の核酸に結合し、標準曲線から細胞数を算出できます。
DNA-to-RNA比は細胞周期の間に変化し得るため、CyQUANTキットでは、RNaseで切断した細胞溶解物と核酸標準曲線を用いて細胞数を決定することができます。
ここでは、CyQUANT キット、SpectraMaxマイクロプレートリーダー、SoftMax® Proソフトウェアの使い方についてご紹介します。2つの方法を詳しく説明します。1つ目の方法では、細胞ベースの標準曲線を用いて細胞増殖を定量します。2つ目の方法では、RNase で処理した細胞試料とDNA 標準曲線を用いて細胞増殖を定量します。
アプリケーションノートをダウンロードする▶
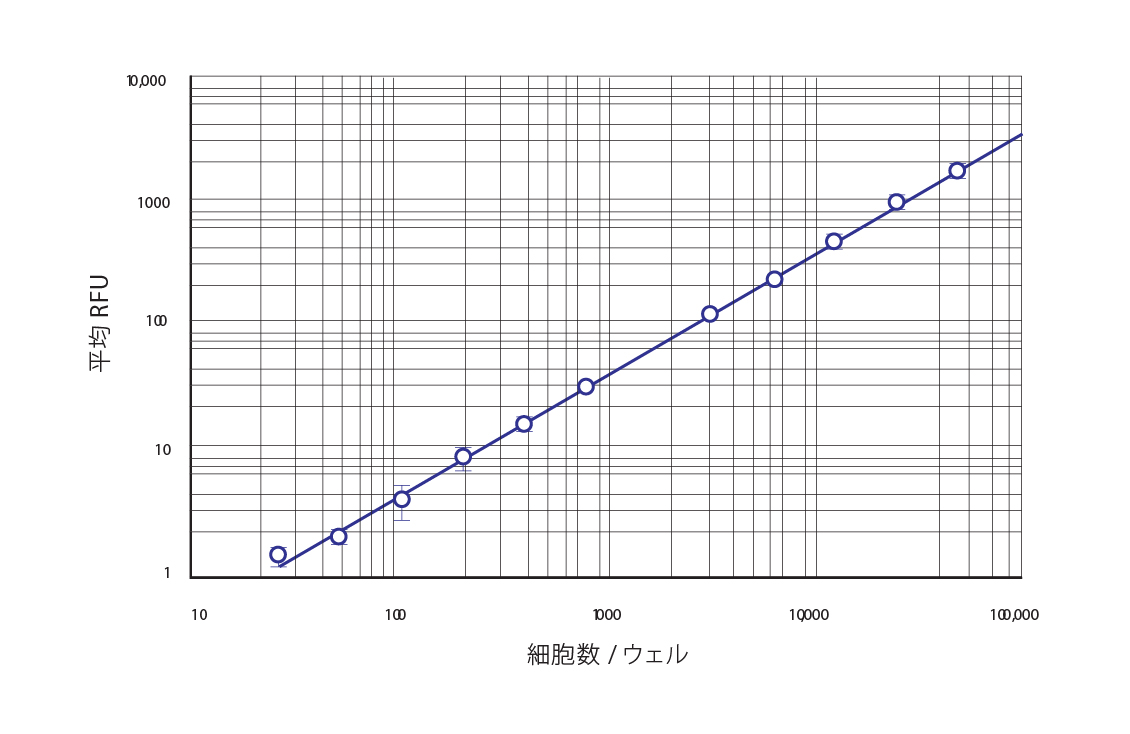
細胞ベースの標準曲線。細胞密度(1ウェルあたり25 ~ 50,000個の細胞)をアッセイした。
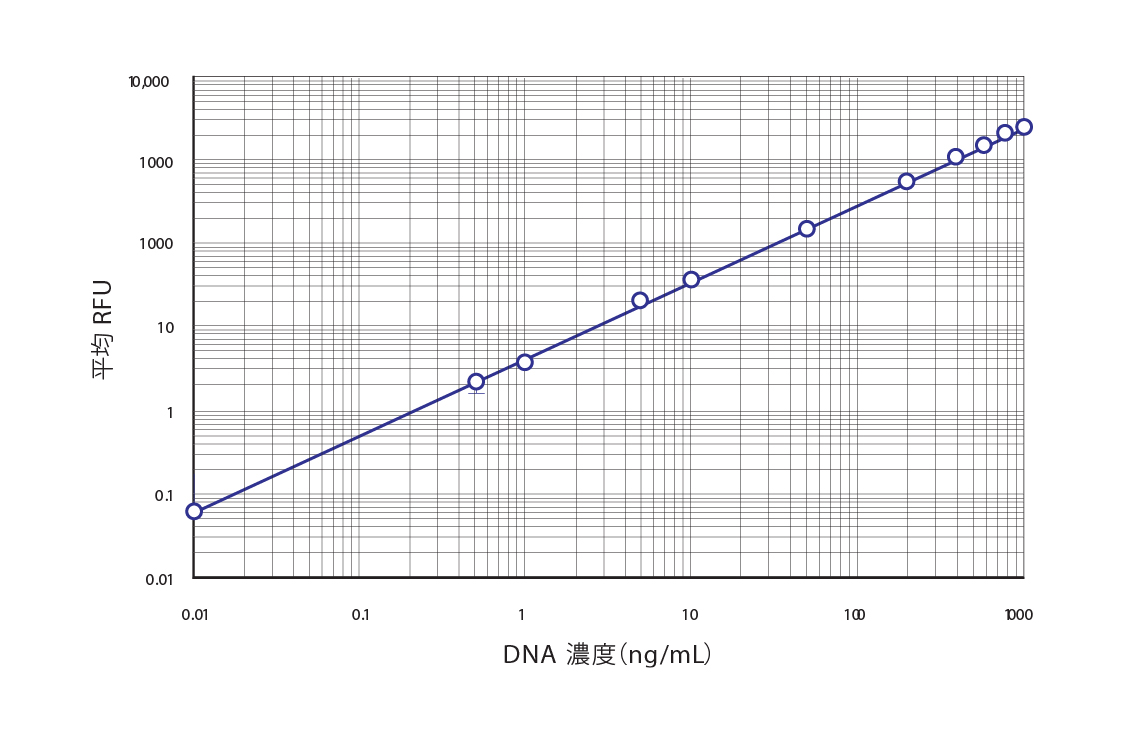
DNA 標準曲線。CyQUANT アッセイキットを用いて作成したDNA 標準曲線。
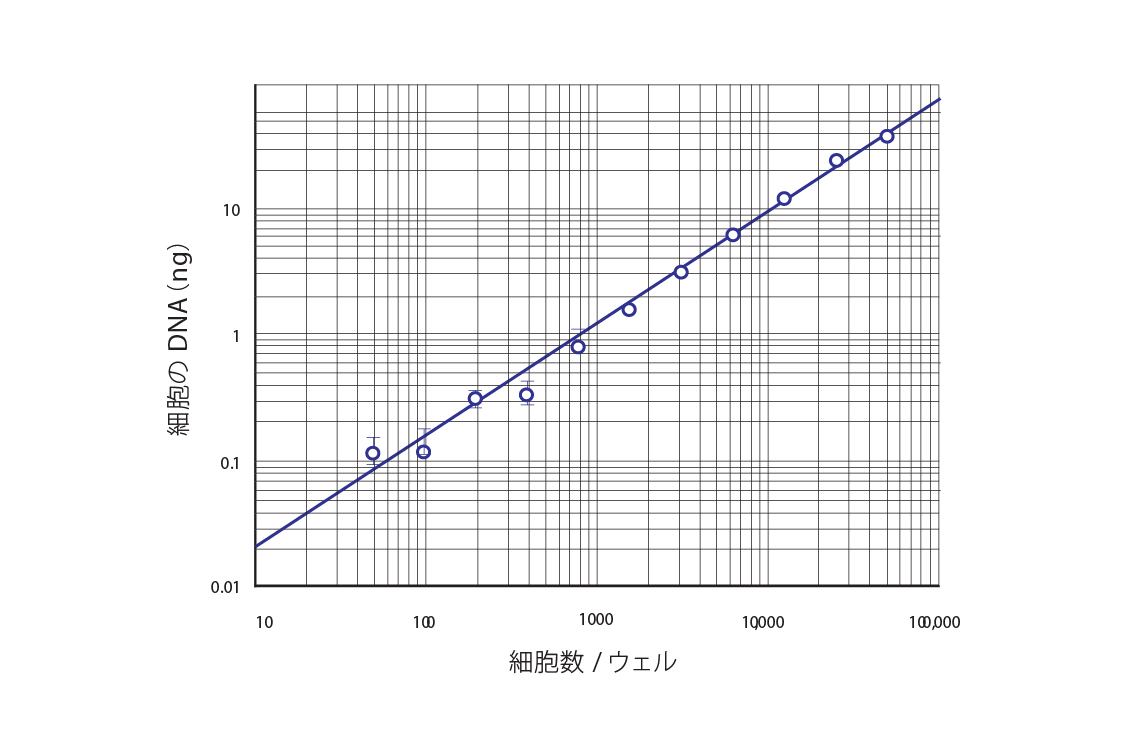
細胞のDNAvs.1ウェルあたりの細胞数。細胞のDNA濃度vs.1ウェルあたりのRNase処理細胞数。
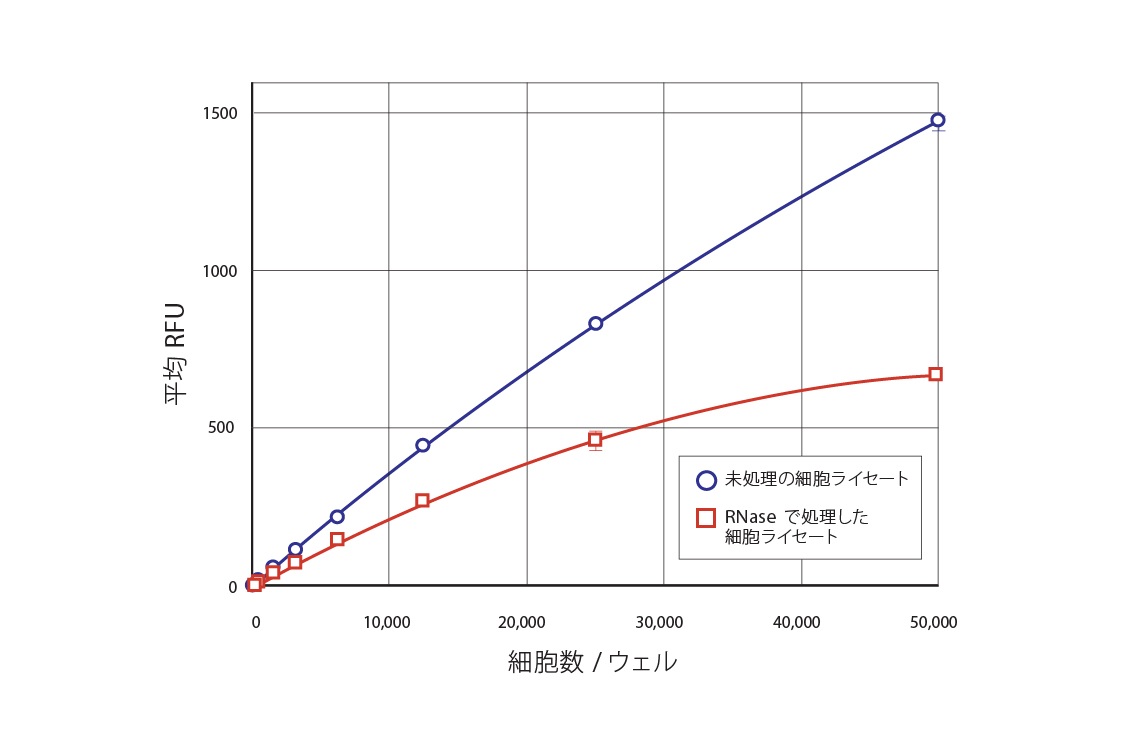
RNase処理および未処理細胞の比較。未処理細胞と比較したRNase処理細胞のRFUvs.細胞数/ウェル。青丸:未処理細胞溶解物、赤四角:RNase 処理細胞ライセート。
- 迅速で簡便な細胞増殖定量
- わずか50 個の細胞を高感度に検出
- SoftMax Proソフトウェアのプリセットプロトコルによる簡単なデータ解析
1cm光路長に自動補正した光学密度測定
UV/VIS分光光度計とマイクロプレートリーダーのビーム構造は、基本的に異なっています。分光光度計では、キュベットやチューブを介して水平方向の(横断的)光路で試料を測定します。水平方向の光ビームと慣例的に使用されている1 cm の光路長により、吸光係数に基づくアッセイを簡単に実施でき、研究室間で容易に結果を比較できます。マイクロプレートリーダーでは、光ビームが垂直方向の場合、光路長が各ウェルの液量に依存します。
マイクロプレートでは光路長がさまざまであるため、吸光に基づくアッセイを行うのは困難であり、マイクロプレートリーダーで得られた結果と分光光度計で得られた結果を比較するのは混乱を招きます。この問題は、SpectraMax® マイクロプレートリーダーとPathCheck® 特性(マイクロプレートの各ウェルの光路長を測定でき、吸光度を1-cm の光路長に自動的に標準化できる機能)の導入により解消されます。このアプリケーションノートでは、PathCheck の基本原理をご紹介し、SpectraMax マイクロプレートリーダーとSoftMax® Pro ソフトウェアでPathCheck を使用する際の具体的な手順について説明します。
アプリケーションノートをダウンロードする▶
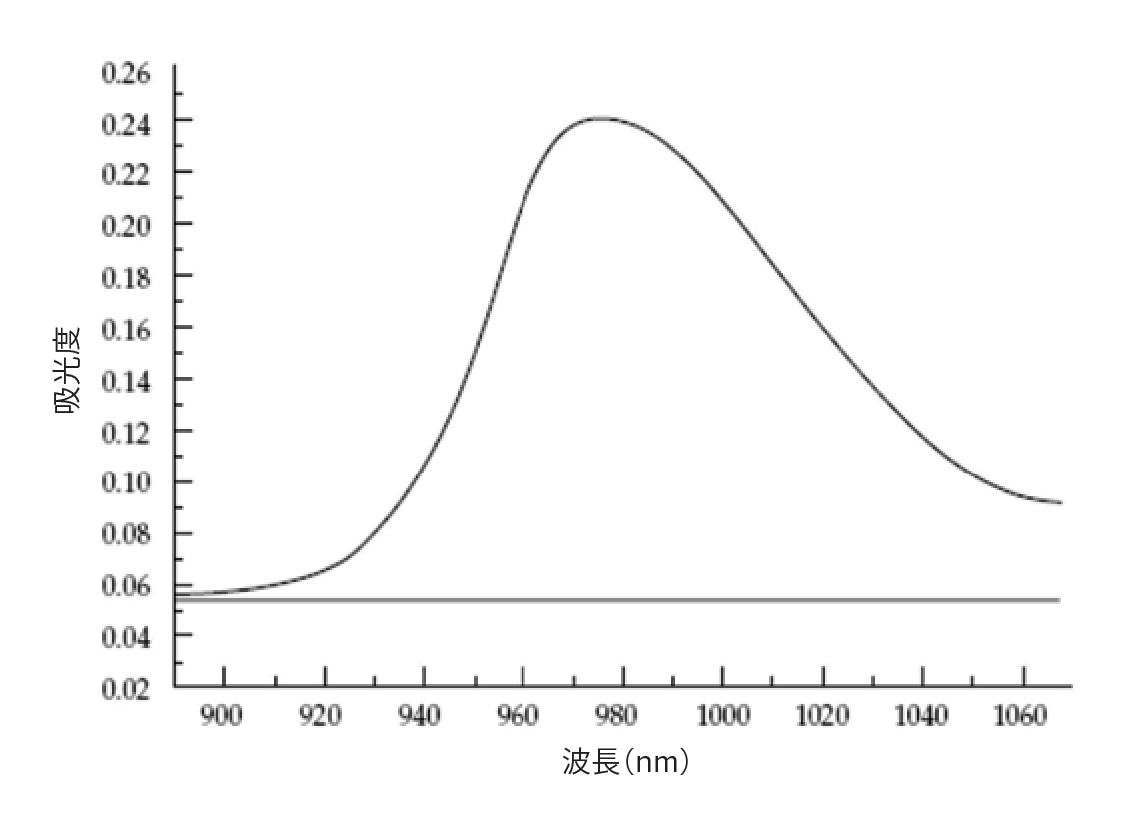
水の吸光度スペクトル。977 nm に吸光度ピークのある水の吸光度スペクトル。
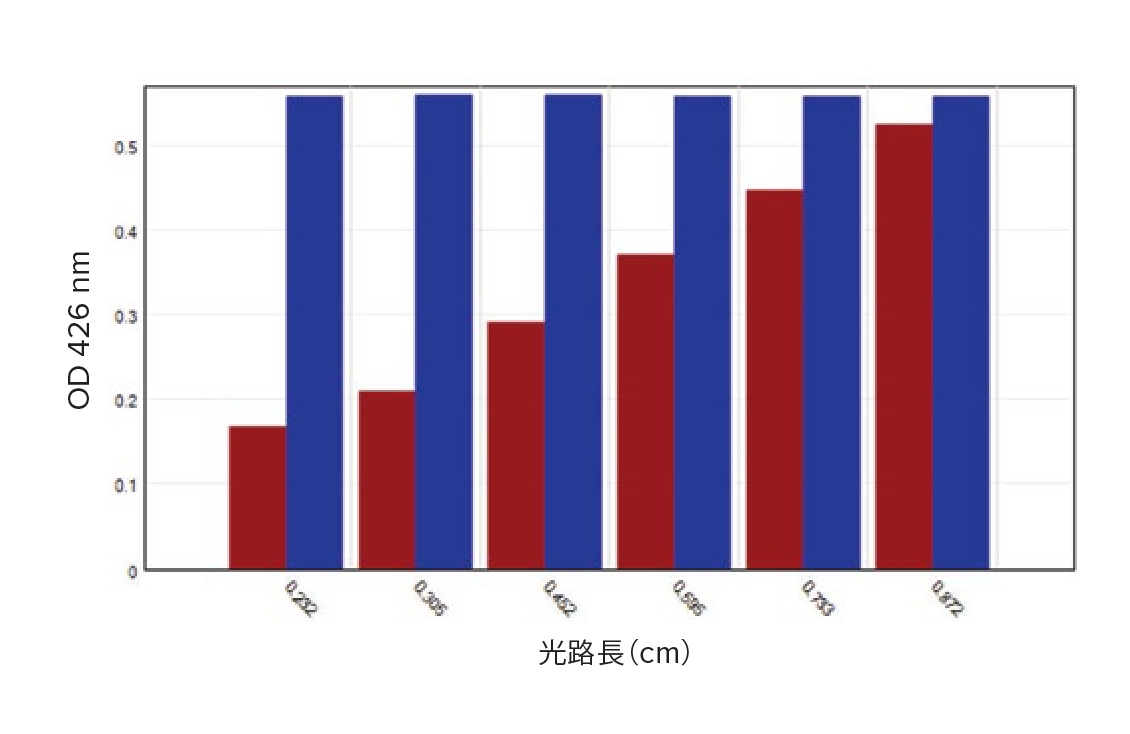
光路長補正ありおよび補正なしのOD426。Red bars:光路長補正なしのOD値、blue bars:光路長で補正したOD 値。y軸に75 ~ 300μLの試料の平均光路長を示す。
- 正確な結果を得るため、さまざまなマイクロプレートウェル容量を自動補正
- 標準曲線不要の吸光度に基づく定量
- 温度非依存性の光路長補正
NanoOrange アッセイを用いたタンパク質定量
吸光度280、BCA、Bradford またはLowryアッセイなどの通常の光度計法は、マイクロプレートフォーマットではあまり感度が高くありません。マイクロプレートフォーマットでのLife TechnologiesのNanoOrange®タンパク質定量キットのダイナミックレンジは10ng/mL ~ 10μg/mLです。このアプリケーションノートで示されたデータからダイナミックレンジと検出下限を確認しました。
NanoOrangeアッセイは、Gemini XPSマイクロプレートリーダーまたはその他のSpectraMax 蛍光マイクロプレートリーダーと、SoftMax® Proソフトウェアを用いることで、タンパク質の迅速かつ高感度な検出法になります。SoftMax® Proソフトウェアの解析能力により、読みやすく、ユーザーがカスタマイズしやすいレポート形式で、定量化を実現します。
アプリケーションノートをダウンロードする▶
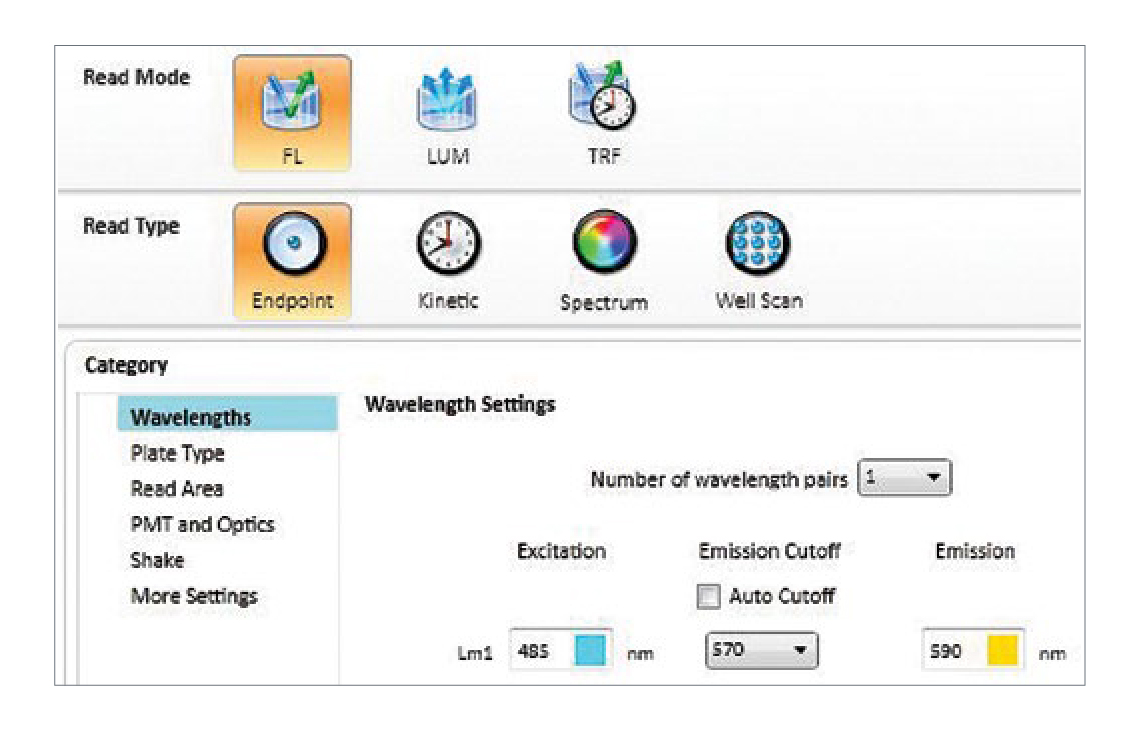
NanoOrangeアッセイ用のプレートリーダーの設定。蛍光検出モードのSpectraMaxリーダーの代表的な設定を示す。
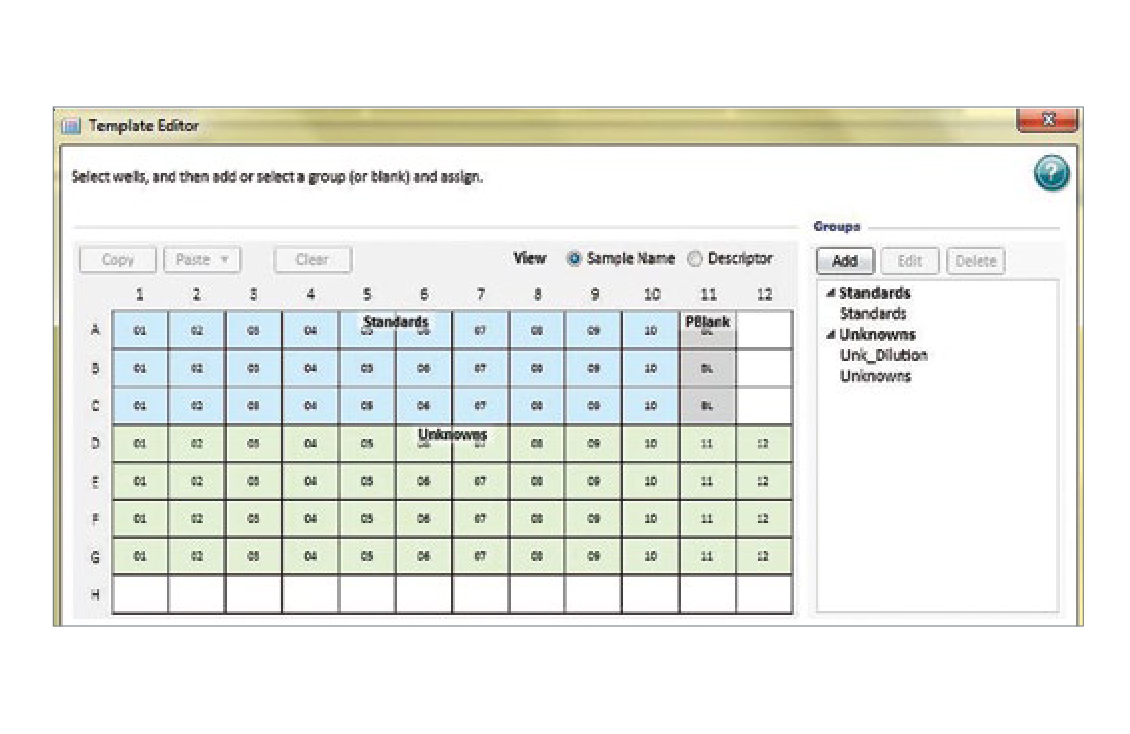
テンプレートセットアップ。Template editorで、ウェルを「Standards」群と「Unknowns」群に割り当てると、標準曲線が自動的にプロットされデータ解析が行われる。
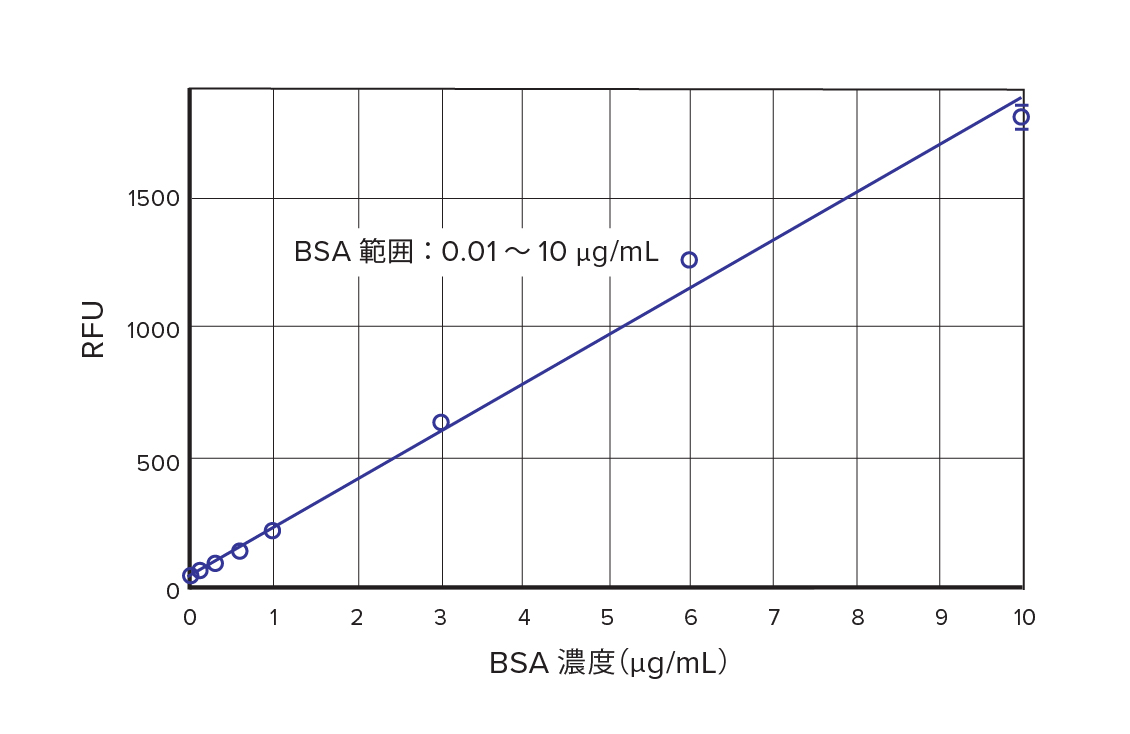
NanoOrange 標準曲線。BSA標準品と線形カーブフィットを用いて得られた標準曲線。
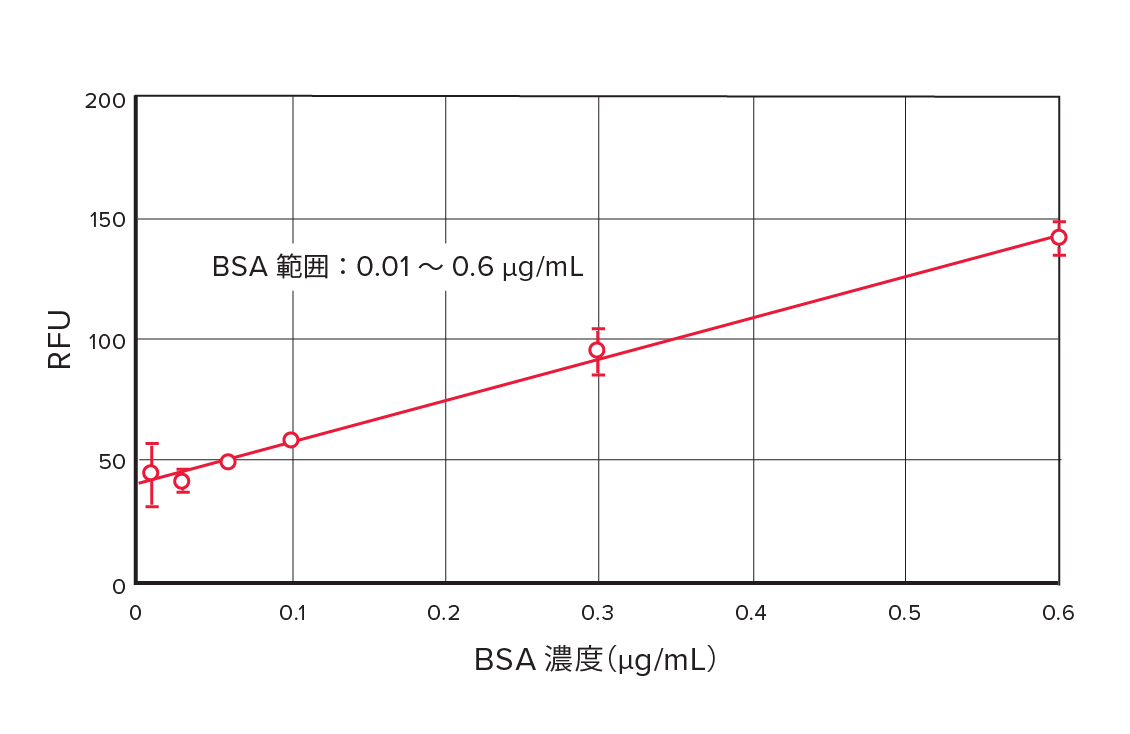
標準曲線下端部の詳細。左図に示した標準曲線の下端部の詳細を示す。
- 吸光法に比べて感度が大幅に向上
- 10ng/mL ~ 10μg/mL の広いダイナミックレンジ
- SoftMax Proソフトウェアのプリセットプロトコルによる簡単なデータ解析
Bradford およびELISA アッセイを用いたタンパク質定量
吸光度は多くのアプリケーションに適した検出法のため、エンドポイントリーダー は、研究室で多くみられます。例としては、サイトカイン定量用ELISA やBradford アッセイを用いたタンパク質濃度の測定などがあります。ここでは、Bradford タンパク質定量アッセイを用いてMolecular Devices EMax® Plus マイクロプレートリーダーとVMaxリーダーの性能を比較します。
また、マウス/ ラットIL-22 の定量用サンドウィッチELISA を用いて、新しいEMax Plus マイクロプレートリーダーとEMax Endpoint リーダーの性能比較も行います。
アプリケーションノートをダウンロードする▶
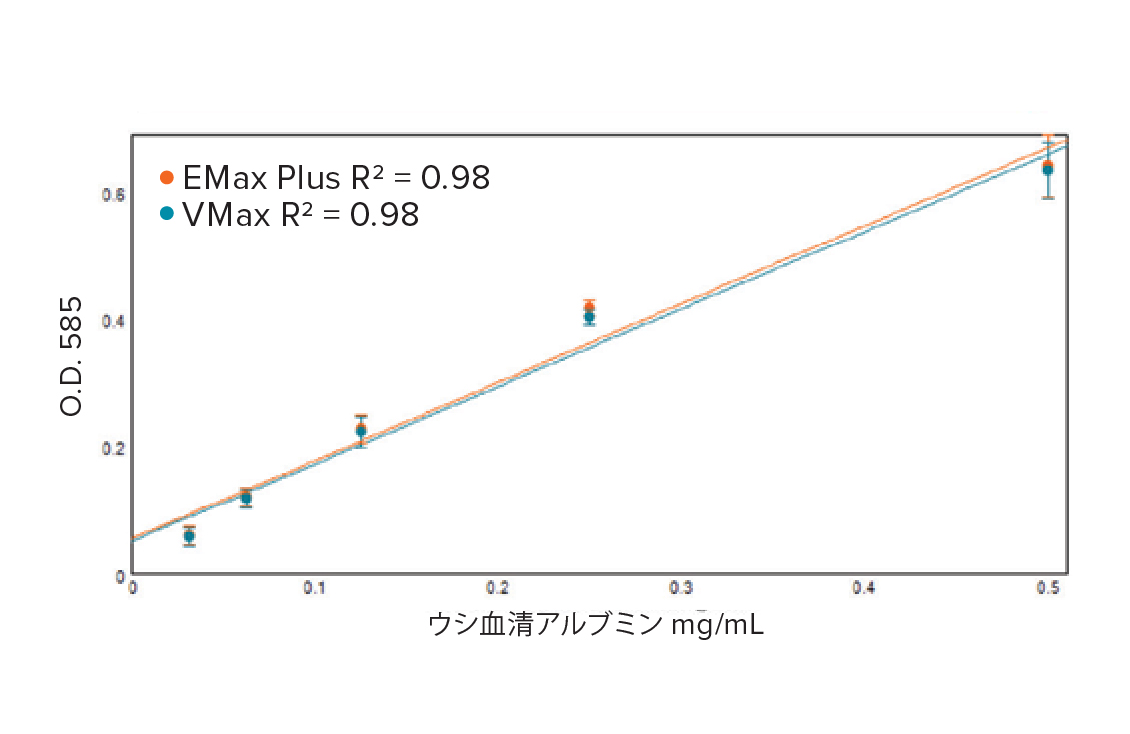
Bradford タンパク質アッセイを用いた吸光度リーダーの比較。Bio-Rad Bradfordタンパク質アッセイを用いて、標準曲線でウシ血清アルブミンの濃度を決定した。2つの機器のシグナル比較はほぼ同一であった。アッセイには5段階の希釈で直線性が認められ、R2値 = 0.98であった。
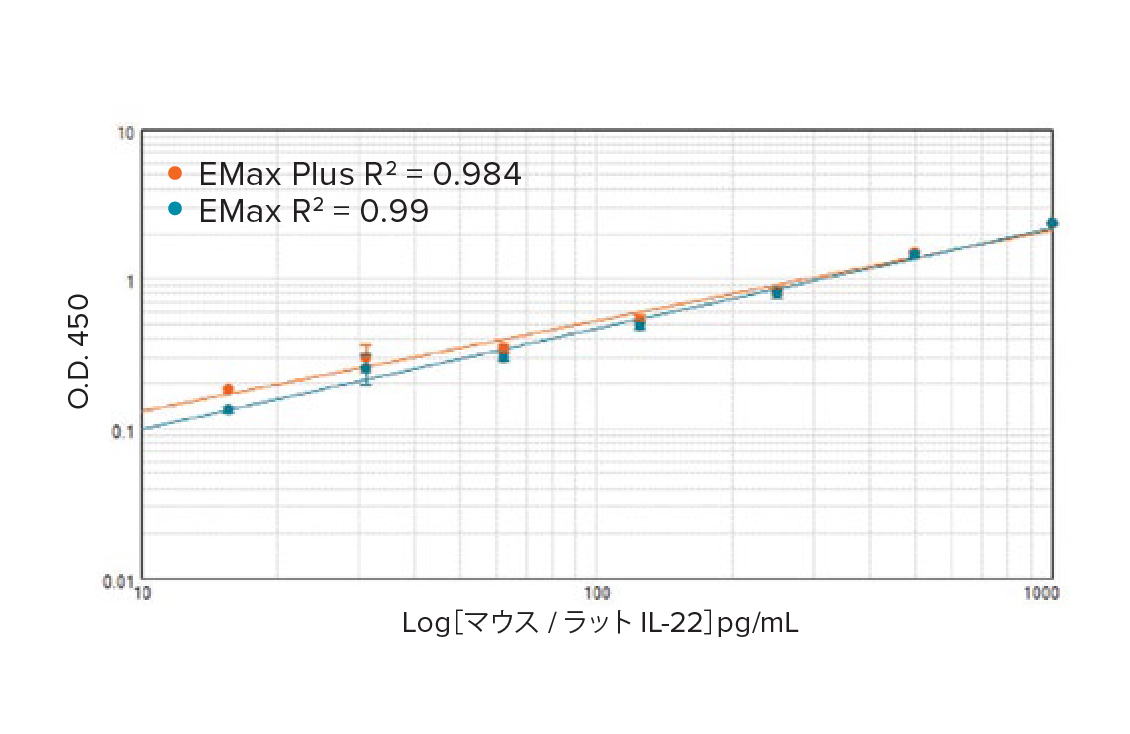
ELISA アッセイを用いた吸光度リーダーの比較。R&D Systemsのマウス/ラットIL-22 Quantikine ELISAを用いて、EMax PlusリーダーとEMax Endpoint リーダーの吸光度アッセイの性能を比較した。IL-22 標準曲線を作成し、ウェルの洗浄にはストリップモードのMultiWash+ Plate Washerを用いて、サンドウィッチELISA を行った。両方のリーダーでELISA プレートを測定後、それぞれの標準曲線はほぼ同一であった。
- 幅広いアプリケーションに対応するため、8 個のフィルターを標準装備
- コンパクトな設置面積
- 直感的な使いやすさ
- SoftMax Proソフトウェアのプリセットプロトコル
少量のDNA 試料の蛍光ベースの定量
UV吸光度を用いたDNAの測定は、多くのタイプの試料を定量できる一般的な方法です。しかし、一部のアプリケーションでは、調製法や原試料の希少さのために、非常に少量または低濃度の試料を定量する必要があります。このようなアプリケーションには、蛍光DNA定量法を用いることにより、高い感度が得られ、低濃度の少量の試料の使用が可能になります。
SpectraMax® マイクロプレートリーダー用のSpectraDrop™ Micro-Volume マイクロプレートは、1 プレートあたり最高64個の試料を処理でき、他の低容量マイクロプレートよりも高い処理量を提供します。わずか2 μL の試料を測定できます。
SpectraDrop マイクロプレートには、特別にデザインされたアダプターとスライドペアが組み込まれており、その光学的透明度により、アプリケーションのニーズに合った吸光度および蛍光モードで測定することができます。
動画を見る▶
アプリケーションノートをダウンロードする▶
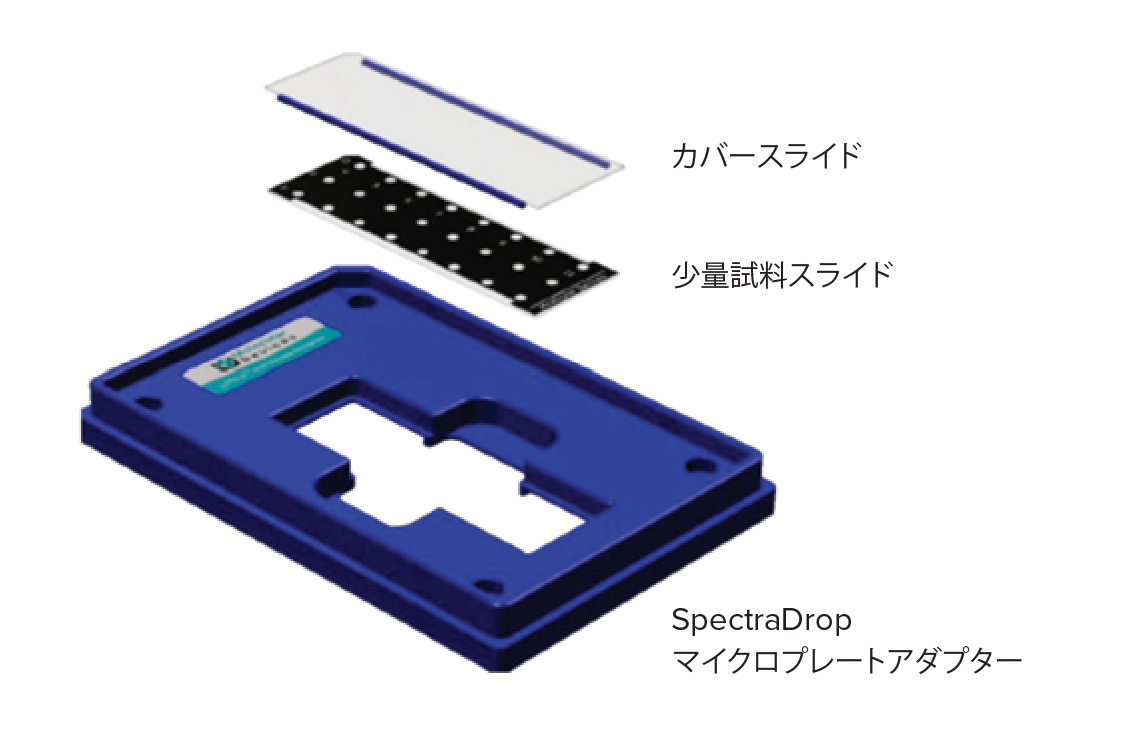
SpectraDrop マイクロプレートの構成。少量試料スライドには、24 個または64 個の試料を保持するスポットまたは「ウェル」を区切るマスクがある。
カバースライドにはそれぞれ2 μL または4 μL 試料で使用するための0.5mmまたは1.0 mmのスペーサーが付いている。
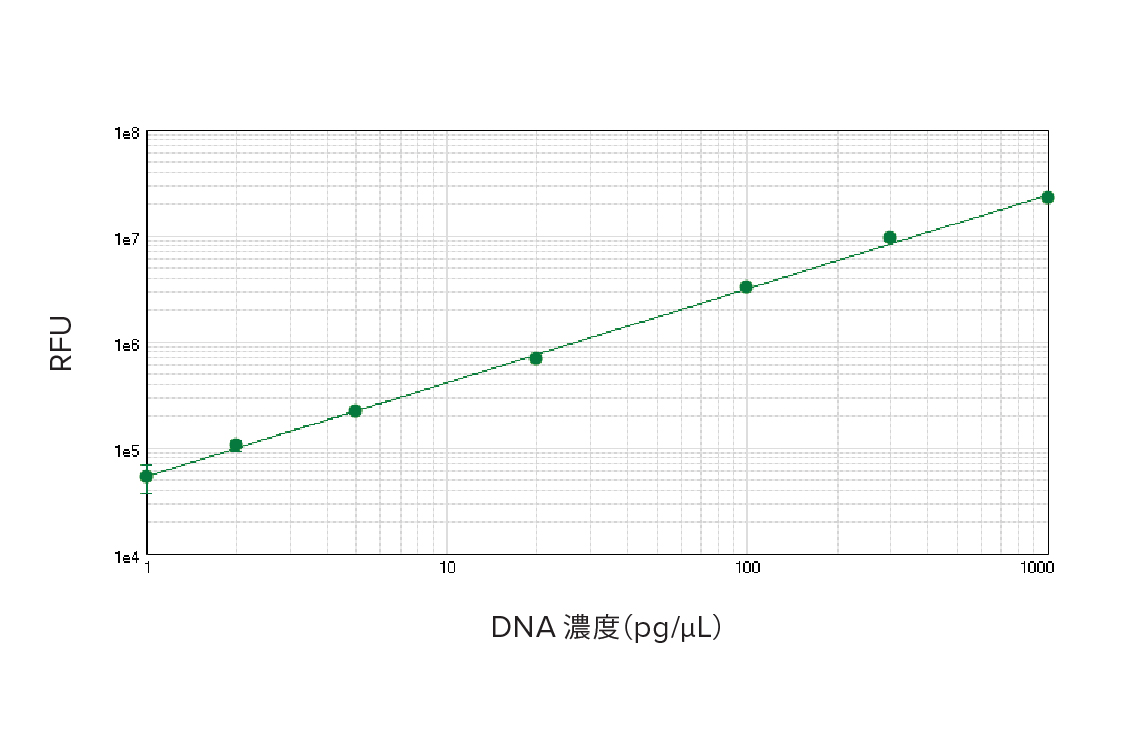
dsDNA標準曲線。SpectraMax Paradigm リーダーに搭載したSpectraDrop MicroVolume マイクロプレートを用いたdsDNA 標準曲線。
dsDNA の検出下限は5 pg/ ウェル、または2.5 pg/μL。標準曲線r2=0.999。SpectraMax M5e リーダーでも同様な結果が得られた。
- 最低2.5 pg/μL のDNA を検出
- 圧倒的な処理能力
- 較正不要のセットアップ
- 洗浄しやすいデザイン




